
背景
据统计类药库含有超过10^60个分子。目前没有任何可用的技术能够筛选出此规模的分子库。因此,基于片段的药物设计(FBDD)自20世纪90年代推出以来,便已成为制药行业的一种主导的药物发现方法。基于片段的方法的主要优点:(1)片段空间可以比类似药物空间更有效地采样;(2)片段的复杂性相对较低, 有助于药物化学家进行结构优化,从而提高了筛选的效率和成功率。
以前的研究也讨论过配体的重要性,由于片段相对较小,片段间的结合较简单,优化过程相对于大型配体具有优势,反观大型配体,它的关键相互作用基团并不容易与不重要的官能团区分开来。但与此同时,由于片段的官能团数量有限,与类药物分子相比,片段的绝对结合亲和力较低,结合往往是杂乱的,因此片段识别命中仍然是项技术挑战。
FBDD作为一种药物发现的方法,主要用于基于生物物理技术来测量蛋白质-配体结合亲和力。在过去的20年里,为将FBDD方法发展成为学术界和工业界的宝贵工具,科学家已经做出了许多努力。目前用于检测片段结合的生物物理技术包括核磁共振(NMR)光谱、表面等离子体共振(SPR)、等温滴定量热法(ITC)和X射线晶体学。这些方法中的每一种都有自己的优点和缺点,因此它们经常在片段筛选中联合使用。虚拟筛选(VS)作为药物发现的另一种方法,已成为一种公认的非常有效的方法。其中包括片段对接和基于形状的相似性筛选。相比之下,基于片段的VS使用频率较低。如前所述,片段的小尺寸和相对简单的结构对区分是否具有亲和力造成了困扰。亲和力的微小差异是VS的主要障碍,其中打分函数不准确、能量近似和有限的构象抽样等也是造成困难的因素。此外,虽然已经发表了许多关于不同药物尺寸的小分子对接的比较研究,但很少有研究来描述基于VS的标准且定义明确的片段筛选实验数据集的有效性,也很难对基于片段的对接工具进行任何有意义的评估。如上所述,FBDD的优点之一是能够更有效地探索化学空间。但实际片段空间远大于通过生物物理技术实际验证的化合物数量。因此,本文利用现有的生物物理技术提出了对这些片段库进行筛选的一个有效且低成本的替代方法SEED,从而作为一个有价值的技术补充基于片段的实验技术的缺陷。
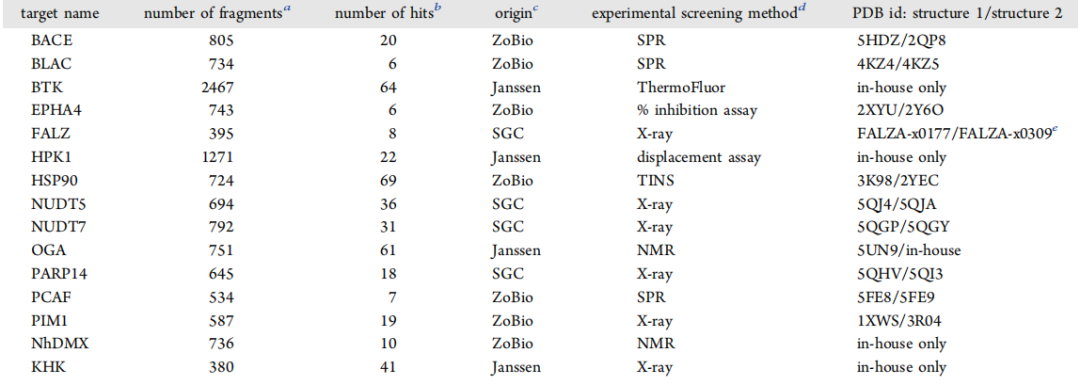
表1 本研究包含的15个数据集
图片来源 JCIM
片段对接程序SEED
SEED是一种开放源程序,主要用于对离散三维空间的精细搜索来对接刚性小分子。SEED源代码在https://gitlab.com/CaflischLab/SEED。该方法于1999年首次发表。SEED中使用的基于力场的势能函数包含四个部分:蛋白-片段之间的静电、范德华、蛋白质去溶剂化以及片段去溶剂化。此外提出了一种计算静电对自由能贡献的广义玻恩方程的有效数值方法,因此片段的对接大约需要1−10秒。
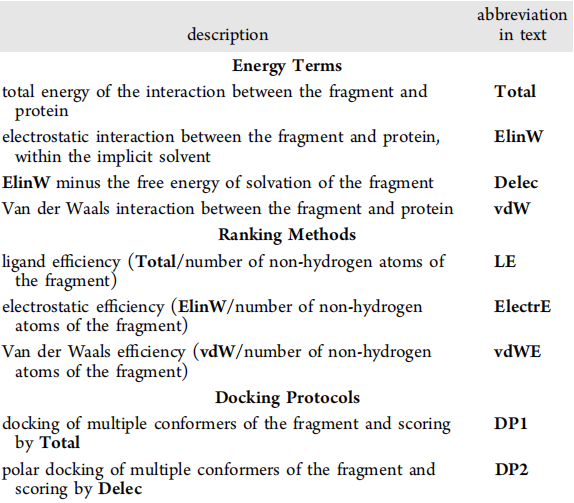
表2 SEED各能量项及两种对接方法(DP1和DP2)的描述
图片来源 JCIM
SEED已成功地用于几个片段筛选,命中率最高可达40%。除了SEED之外,很少有专门用于片段对接的工具。LUDI是基于规则的对接程序,是唯一的除SEED外用于片段对接的工具。传统的对接程序,如Flexx,GOLD,DOCK,Glide,Autodock和AutoDockVINA也适用于片段对接,但性能评估很大程度上是不存在的。本文进行了一项回顾性对接研究来分析SEED在15个不同的蛋白质靶点上的对接性能。此外,还与Glide程序进行了比较,Glide是一种公认的对接工具。库的大小从近400到3500个片段不等,实验命中率从0.8到10.8%。评估了SEED对接的潜力,讨论了SEED中基于片段筛选的最优协议,并提出了一个通用的操作流程。
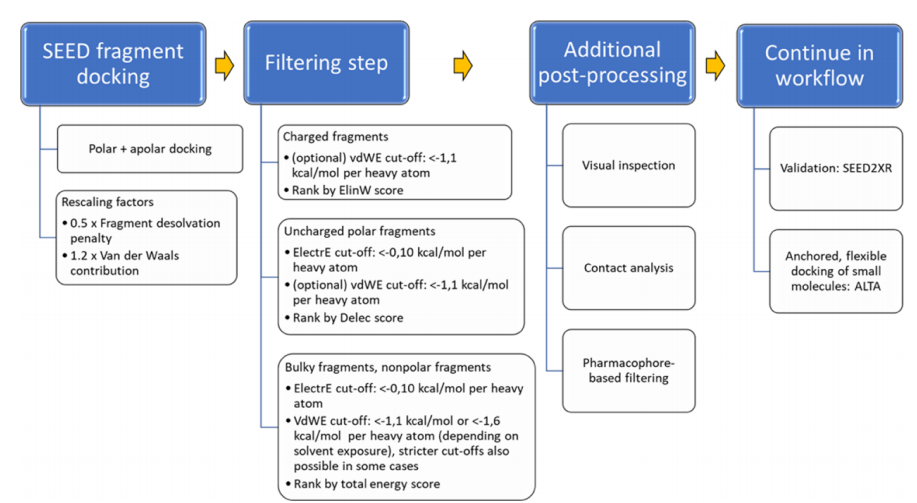
图1 SEED片段筛选的工作流程
图片来源 JCIM
实验方法
首先进行蛋白质结构和片段库的制备。蛋白质结构的RMSD会对VS结果产生很大的影响,为探究这一影响,本文为每个数据集制备了两个不同的蛋白质结构,以考虑结合位点的微小构象变化对VS的影响。选择的结合位点是位于全息蛋白晶体结构中配体或片段数量最多的位点。其次,利用精确抽样优化了酰胺(Asn和Gln)、羟基(Ser、Thr和Tyr)和硫醇(Cys)基团和His残基的互变异构状态。片段库作为SMILES导入到MOE中,并转换为二维结构。由于SEED使用配体的刚性构象进行对接,除了FALZ、NUDT5、NUDT7和PARP14数据集外,还排除了具有两个以上可旋转键的分子。对于这四组,筛选出具有两个以上可旋转键的片段生成包含300个片段的库。然后以SDF格式将库导入Maestro,并使用Lig Prep模块为pH7±1处的所有发生的原生质体生成三维(3D)构象,保持库中定义的互变异构形式。然后,使用ConfGen生成多达64个构象,用于与SEED对接每个片段。接下来进行数据集的分析和片段的对接,采用SEED程序进行对接。程序SEED中结合能的评估由基于力场的能量函数组成,蛋白质和片段中原子的部分电荷和VDW参数分别取自CHARMM36全原子力场和CHARMM一般力场。
实验结果
对SEED性能的判断主要是基于筛选能力,即通过评分函数区分活性和非活性化合物的能力。下图为对SEED方法的评估结果。
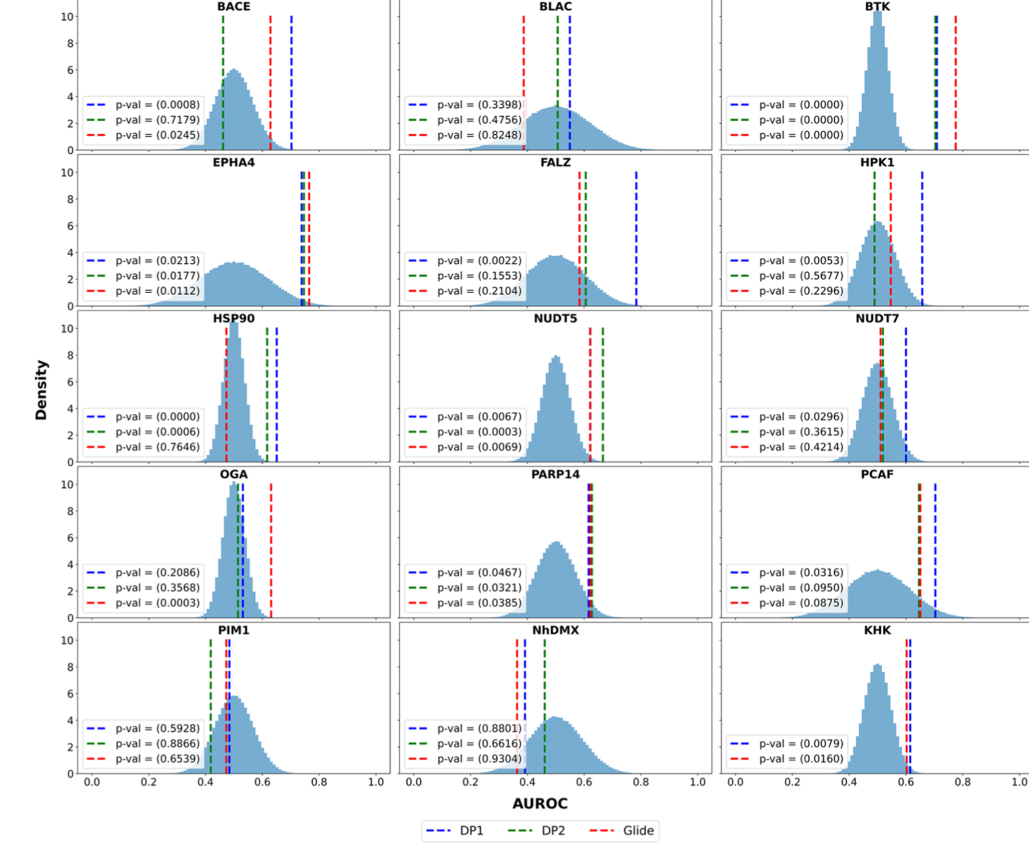
图2 不同对接方法的排名和AUROCs的直方图
图片来源 JCIM
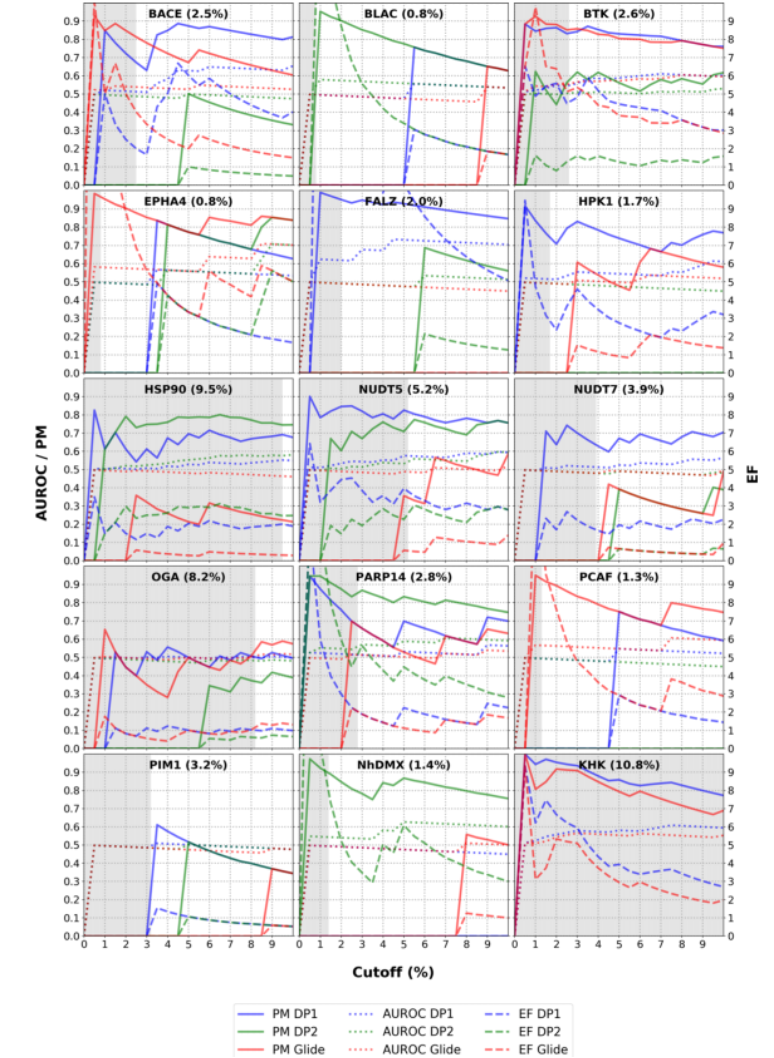
图3 SEED(DP1和DP2)与Glide的性能对比
图片来源 JCIM
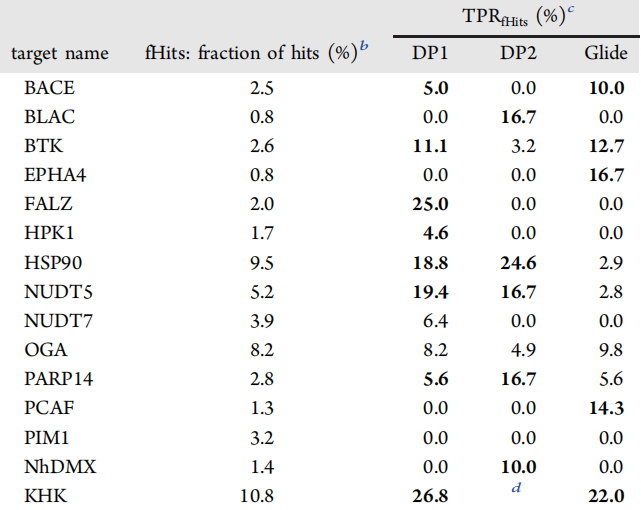
表3 三种对接方法之间的比较
图片来源 JCIM
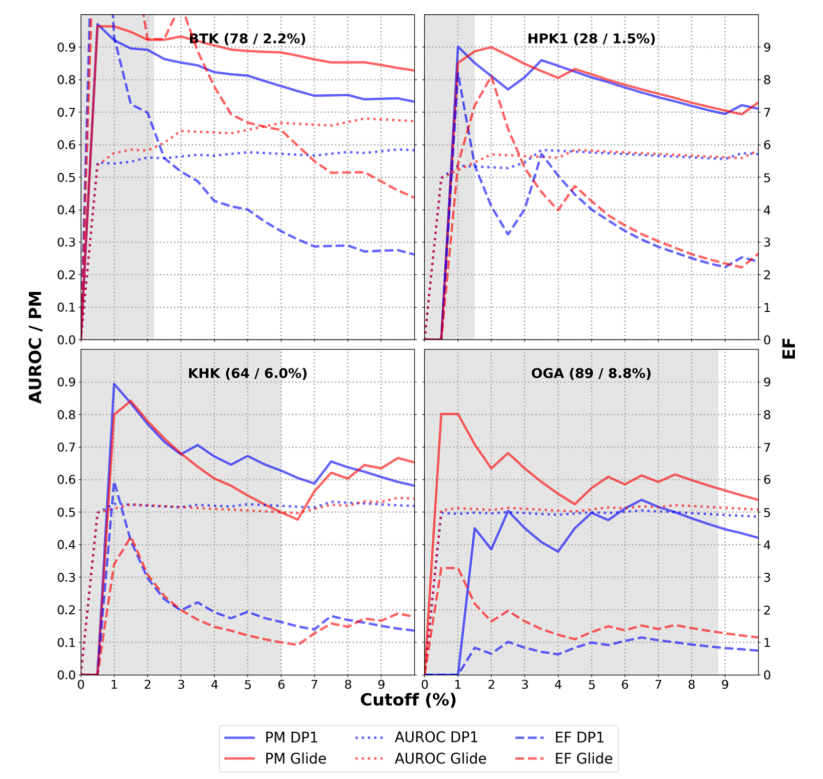
图4 SEED性能与GLIDE的对比
图片来源 JCIM
讨论
1. 数据集:片段对接数据集的公共可用性较低。
2. SEED与Glide相比,SEED在筛选小片段方面一般优于Glide。
3. 定义结合位点:最大的问题是配体结合时构象可能发生变化。
4. SEED和带电片段:目前已知的SEED评分函数问题,以及基于力场的方法的一般问题,是很难找到一个统一的评分标准,适用于带不同形式电荷的片段。
5. 用SEED对接柔性片段:当更灵活的片段包含在库中时,SEED的表现略差于Glide,不太适用于大型、柔性分子的虚拟对接。
6. 由于本研究验证了SEED优于随机片段选择和提供早期片段采集的能力,因此没有进行广泛的后处理。
结论
本文评估了高通量对接程序SEED对于筛选片段库的有效性,并得出结论,SEED可用于片段库进行筛选。虽然对于柔性片段对接Glide比SEED更快,但SEED在大多数情况下优于Glide。由于特异性蛋白结合和弱结合引起的不准确,实验片段筛选结果可能不一致,分别产生假阳性和假阴性。此外,片段可以与蛋白质上的替代结合位点结合,导致假阳性的产生。因此,用于虚拟片段筛选的真实EFS可能高于报道值。此外,适当的后处理、调整操作方法和工作流程对虚拟筛选的结果也有重大影响。综上,本文开发了一种高性能的SEED虚拟片段筛选工作流程,它结合了三种不同的方法,分别用于带电、极性和非极性片段。
参考文献
Assessment of the Fragment Docking Program SEED, Kenneth Goossens, Berthold Wroblowski, Cassiano Langini, Herman van Vlijmen, Amedeo Caflisch, and Hans De Winter* J. Chem. Inf. Model. https://dx.doi.org/10.1021/acs.jcim.0c00556