引言
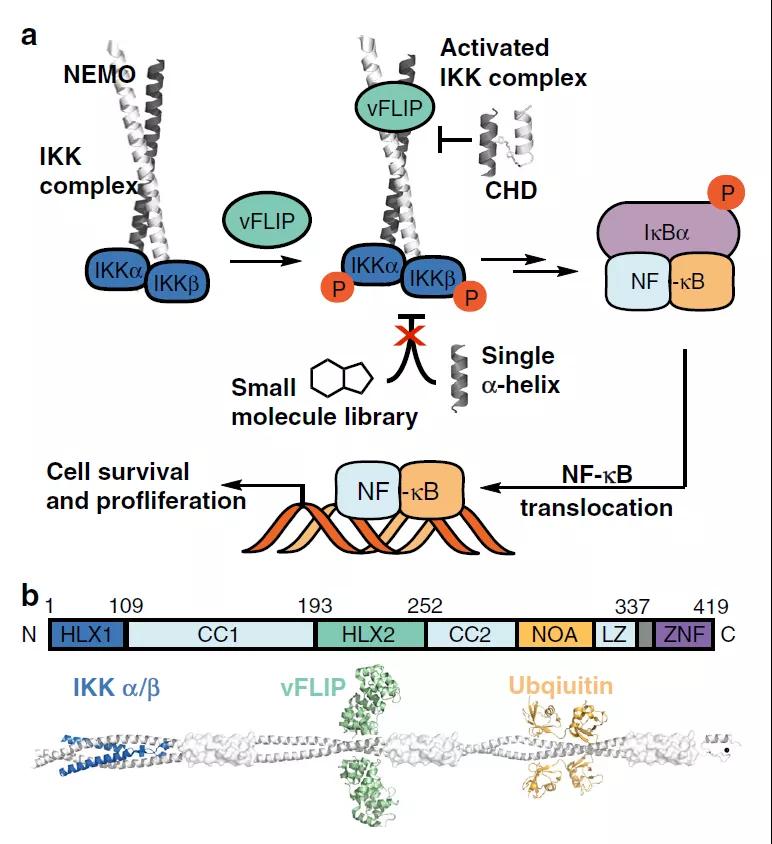
蛋白质-蛋白质相互作用(PPIs)指一种蛋白质对另一种蛋白质的选择性识别,其控制着细胞生命的三个关键维度:生长、存活和分化。这些相互作用的调节对于理解支配生物功能的细胞网络和开发新的治疗方法都是至关重要的。NEMO是NF-κB信号转导中心的支架蛋白,不同的调节因子与NEMO的不同卷曲区域发生相互作用,但该蛋白的拓扑复杂性限制了抑制剂的设计。本文中作者通过小分子筛选和合理设计,全面阻断了卡波西肉瘤疱疹病毒癌蛋白vFLIP与NEMO之间的相互作用。
先睹为快
作者单位
康奈尔大学医学院(Ethel Cesarman & Paramjit S. Arora),纽约大学化学系(张颖凯教授),哈佛大学医学院。
靶点
NEMO-vFLIP:NF-κB必须调节蛋白-病毒FLICE抑制蛋白(PDB ID:3CL3)
筛选数据库
38,506个小分子:LOPAC (Sigma, 1280个), Enamine 3 (33,135个), NIH临床(727个), HTRSC临床(294个), Prestwick (1109个), Pharmakon (905个)
计算方法
结合口袋分析,丙氨酸扫描
计算软件
AlphaSpace软件
计算流程
为发现阻断vFLIP-NEMO蛋白相互作用的有效抑制剂,本文中作者首先采用时间分辨荧光共振能量转移法筛选了38,506个小分子,命中的9个小分子均未表现出特异性。随后作者采用AlphaSpace软件对vFLIP-NEMO晶体复合物结构进行了活性口袋分析,确定PPIs相互作用的关键氨基酸残基主要位于NEMO的螺旋2上,结合丙氨酸扫描分析提供的氨基酸突变建议,最终设计了并合成了抑制剂CHD3NEMO,CHD3NEMO提升了口袋占有率,表现出与vFLIP的高亲和力,活性实验也表明其可有效的阻断vFLIP与NEMO之间的相互作用。

小分子抑制剂的高通量筛选
作者采用基于靶标的方法来发现vFLIP-NEMO相互作用的抑制剂。作者对数据集中的38,506个小分子采用时间分辨荧光共振能量转移(TR-FRET)法筛选vFLIP抑制剂,命中了20个抑制率大于40%的小分子,并用剂量反应曲线进行了体外活性验证,最终发现有9个化合物的IC50值小于65 μM,然而,命中小分子均没有表现出对PEL细胞(BC-3)的特异性毒性,表明非特异性结果与vFLIP独立机制有关。
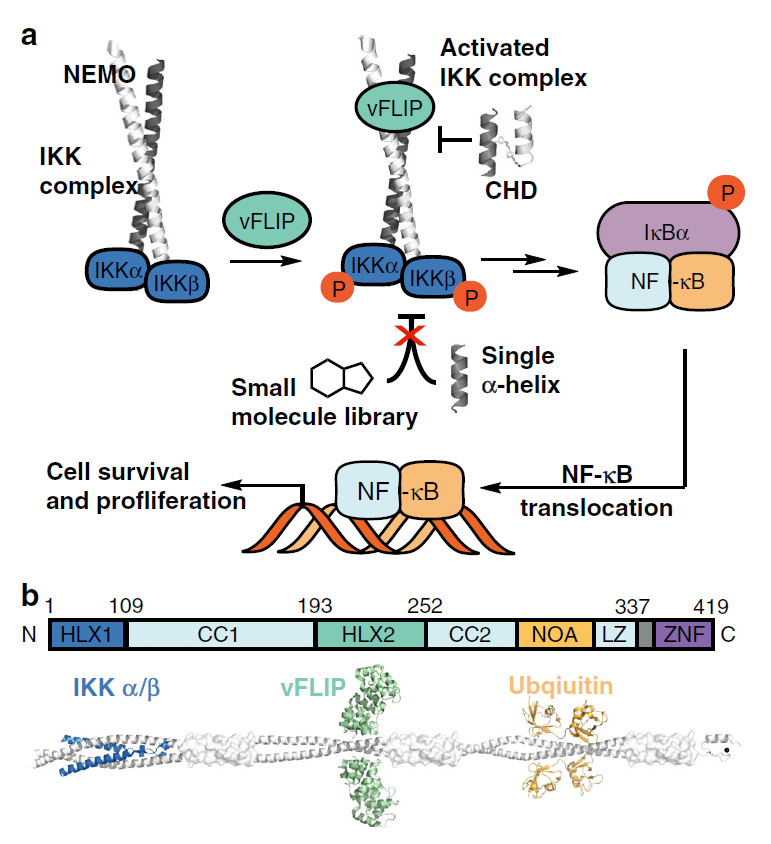
图1. vFLIP介导激活NF-κB 信号通路
图片来源:Nature Communications
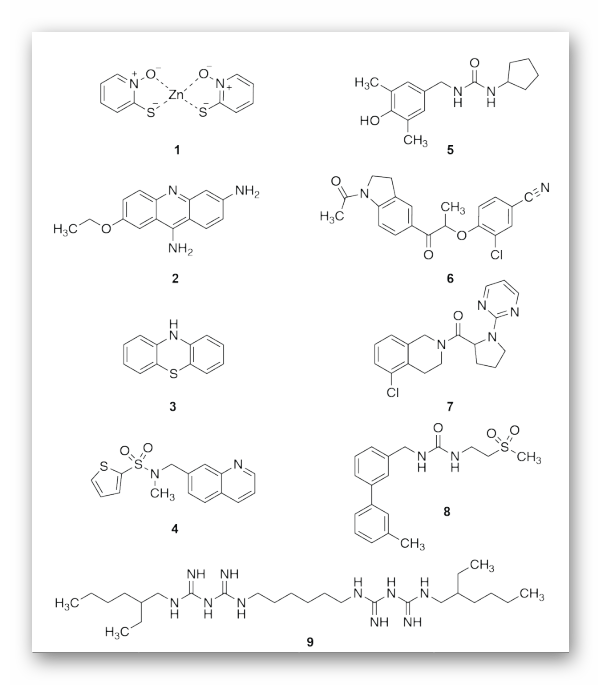
图2. 使用TR-FRET分析筛查的前九个命中小分子
图片来源:Nature Communications
活性口袋的分析
随后,作者转向利用理性设计的方法来开发特定的NEMO/vFLIP抑制剂。为确定PPIs中未被充分利用的口袋,作者从vFLIP-NEMO晶体复合物结构出发,利用AlphaSpace软件来表征残基中心口袋,以设计能够提升口袋占有率、更加有效的NEMO/vFLIP抑制剂。结果产生了6个表面口袋:3个高容量口袋(与Helix 1上的Glu240、Helix 2上的Phe238和Helix 2上的Lys246相关联),2个中等容量口袋(与Helix 1上的Ala233和Helix 2上的Asp242相关联),以及1个低容量口袋(与Helix 2上的His235相关联)。
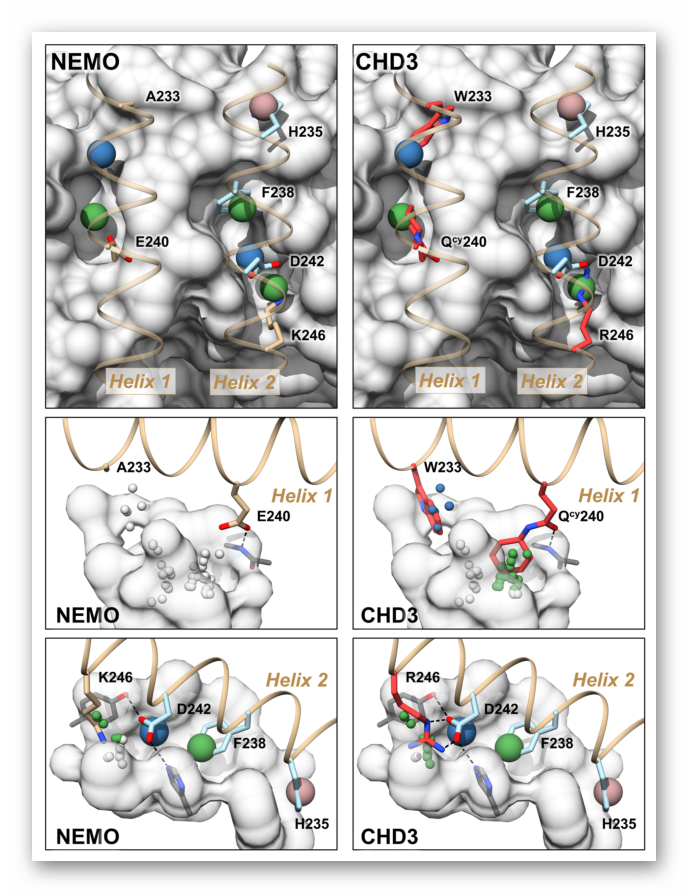
图3. AlphaSpace设计
图片来源:Nature Communications
NEMO卷曲螺旋结构的设计与评价
通过丙氨酸扫描计算揭示了PPIs相互作用的关键氨基酸残基主要位于NEMO的螺旋2(Tyr234,His235,Phe238,Tyr241,Asp242,Ile245)上。基于该结果并结合AlphaSpace软件计算结果所提供的氨基酸突变建议,作者对NEMO卷曲螺旋结构进行了优化设计,如将Lys246突变为Arg246可使口袋IV的占有率从45%增加到75%,为了获得最佳的螺旋间隙堆积,将关键热点残基Tyr234突变为Leu234等。另外,作者设计了非天然的环己胺作为谷氨酰胺衍生物(Qcy),既保留了Glu240与vFLIP口袋衬里残基Phe53骨架之间的氢键,又将环己基延伸到疏水的vFLIP口袋中,具有良好的互补性。作者将突变建议与Qcy都整合到CHD3NEMO中,AlphaSpace预测CHD3NEMO将是vFLIP的高亲和力配体,生物活性及机制研究也证实了该结果。本文所采用的方法是蛋白质-蛋白质相互作用研究的一种有效策略。
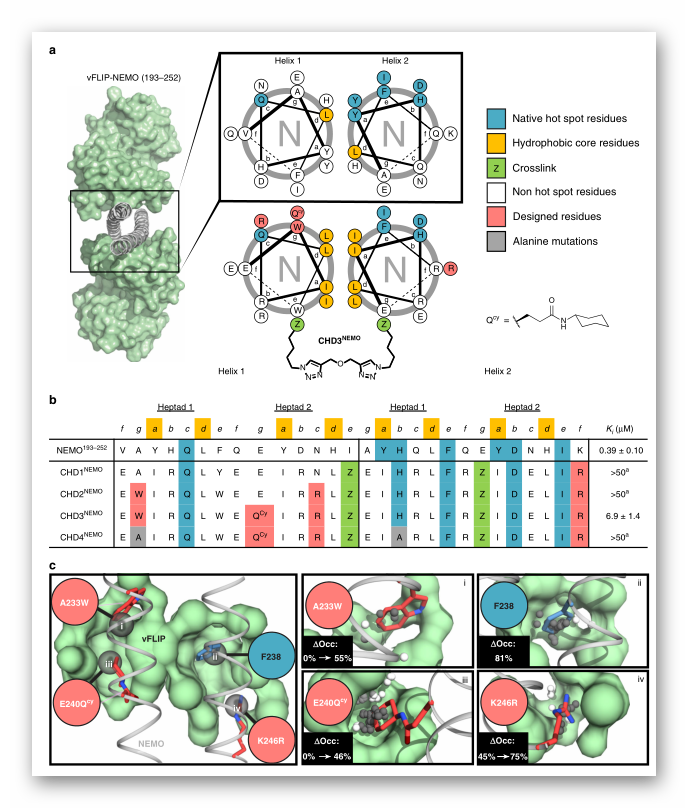
图4. NEMO-vFLIP相互作用抑制剂的合理设计
图片来源:Nature Communications
参考文献
Sadek J, Wuo M G, Rooklin D, et al. Modulation of virus-induced NF-κB signaling by NEMO coiled coil mimics[J]. Nature Communications, 2020, 11(1): 1-14. DOI:10.1038/s41467-020-15576-3