引言
法国创新研究院研究者借鉴计算机视觉方法,提出了一种新的叠合和比较蛋白质空腔计算方法ProCare,有望应用于基于片段的药物设计。
背景介绍
蛋白质的三维结构是基于结构药物设计的基础。潜在空腔可以用大量的计算工具在大分子表面检测,通常可分为三类:基于几何型(如CavBase, VolSite, Fpocket)、基于能量型(如GRID, Q-SiteFinder)和基于进化型(如SURFNET-ConSurf)。基于几何型依赖于对目标分子表面的预先计算来确定可触及的口袋,而基于能量型则依赖于计算目标蛋白和几个探针原子在三维晶格上的相互作用能,最后,基于进化型的工具需要对同一家族的靶点进行多重序列或结构比对,以确定与特定配体结构识别相关的进化保守基序。有趣的是,结构耐药性或配位性,可以通过在已知的可给药和不可给药位点集上训练的机器学习模型,快速计算出来。蛋白质配体结合袋的许多描述符(指纹、距离计数、药效三联、网格点、点云、图形和形状)可以通过几何哈希或小基团检测算法来找到最突出的共享特征,指导蛋白质空腔的结构排列。
结果与讨论
在计算机视觉、模式识别和机器人技术中,点云配准是找到使两个点云对齐的最佳空间变换(如缩放、旋转和平移)的过程(图1)。
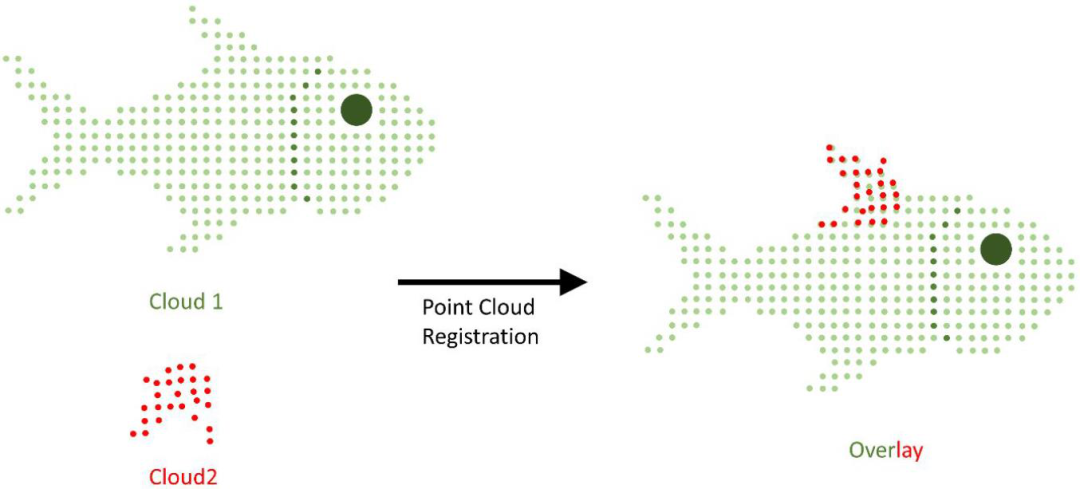
图1 点云配准的示意图表示。将红色云沿其三个主要轴旋转和平移,直到找到与绿色云的最佳对准。
两个点云(cloud 1和cloud 2)配准的基本原理是,首先需要识别对等价点。如果cloud 1和cloud 2中的两个点共享相似的微环境,即相邻点的拓扑布局相似,则认为它们是等效的。因为目标是匹配两个几何形状,所以这里用一个角度值直方图描述一个点的环境,称为快速点特征直方图(FPFH)。
由于FPFH的每个描述符都是一个特定角度值范围的“计数”,两个FPFHs的相似性可以通过一个简单的欧几里得距离来估计。然而,FPFH虽然复杂,但不能避免在检测通信中的歧义,尤其是当存在不应该被考虑的无关点(称为离群值)时。一种排除离群点的方法是随机样本一致(RANSAC)算法。在每次RANSAC迭代中,在cloud 1中随机抽取几个点,分配它们在cloud 2中的对应点,通过比较拓扑距离来验证这些对应点的相关性,最后估计一个旋转/平移来对齐采样集。这种基于少数点的初步校准,然后用迭代最近点(ICP)方法进行细化,它能使两个云中对应点之间的总体均方根偏差最小化。
有趣的是,点云配准很少用于覆盖蛋白质和配体的分子表面。在这里,云被描述为定期填充口袋的3D点的集合,每个点都有一个特定的药效特性(颜色)来补充附近的蛋白质环境。因此,该云更大(200-300点),在形状和药效学特性上具有规律和互补性。我们将首先演示应用这种计算方法来解决蛋白质空腔排列问题的概念证明,接下来对一组参数进行微调,从而在一个已知空腔的大型数据集上实现最佳性能,然后提出一个物理化学相关评分来量化序列和口袋相似度。
ProCare的实现和参数优化。 许多点云配准参数对对准质量有很大影响。因此,研究者通过列举157,465个参数组合,系统地研究了15个关键参数(表1,计算方法),以考虑它们的影响及其相互依赖性。为了测试所有这些条件,研究者设计了一个由五个不同的腔对完成的五个相似的对(EASY1 set)的数据集,只是为了过滤掉那些不能产生任何类型的对齐(fitness = 0),或者不能很好地区分相似和不同对的参数组合(ROC AUCs <1),这两个简单的过滤器可以将潜在组合的数量从157,465减少到20,181(图2)。
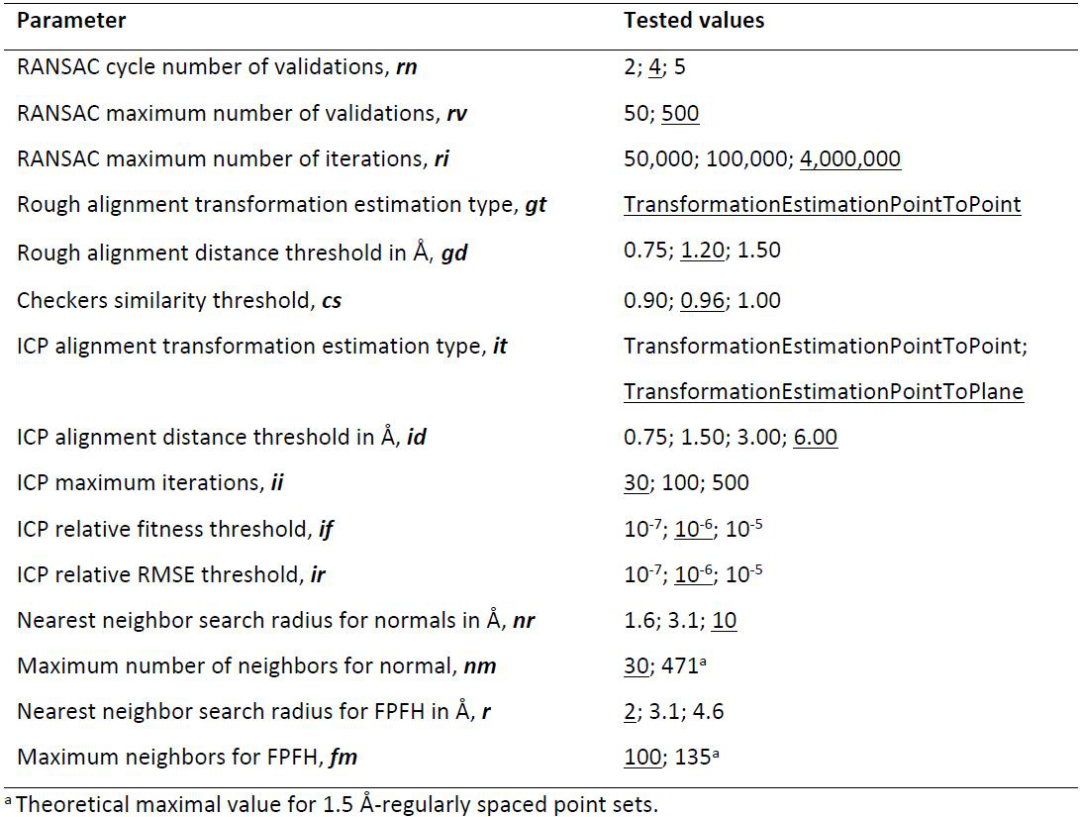
表1 ProCare对齐的Open3D参数值(默认值下划线)
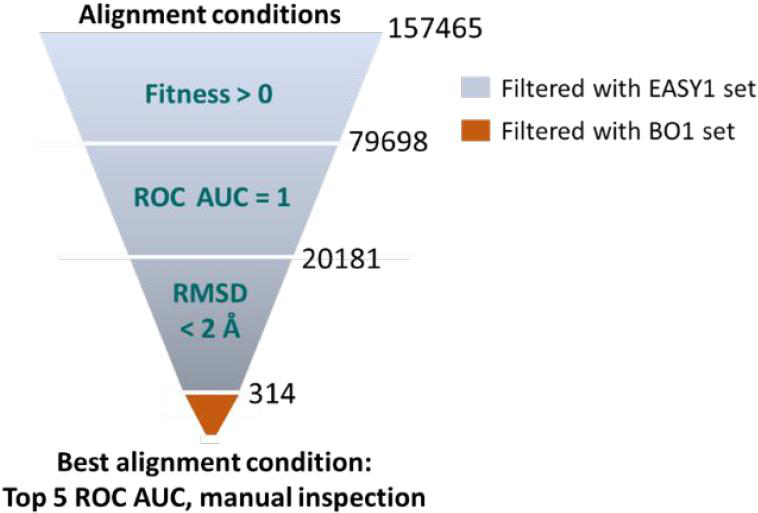
图2 选择程序,确定最佳对准参数。
为了对剩下的314种对齐条件进行基准测试,研究者从16,034个药物-蛋白配体复合物的sc-PDB存档出发,设计了一个更大、更多样的相似对和不同空腔对的数据集(BO1 set)。BO1数据集包括了766对非冗余的VolSite空腔(383相似对,383非相似对),覆盖了507个不同的蛋白(460在相似的集合中,178个在非相似的集合中),62套不同的Uniprot功能注释用于相似对,38套不同的功能注释。
314个预先选择的条件被用来从BO1组中对齐腔对。通过使用三种可能的评分函数(ph4-strict、ph4-rules和ph4-ext)来计算二分类(相似、不同)的ROC曲线下面积(ROC AUC),根据药物学匹配的模糊性对每个条件进行排序。根据ph4-ext评分,研究者最终选择了ROC AUC值为0.87 (CI =[0.85;0.89])的最佳比对条件。使用BO1设置的最优参数集,但使用FPFH-colored-icp方法对粗糙RANSAC比对进行细化,证实了研究者的初始假设,即对应的AUC(ROC AUC = 0.83;CI =[0.81;0.86])低于上述报道。
利用直方图编码、形状和药效特性对方法进行改进。鉴于通过FPFH-icp获得的结果以及出现的不对齐问题,研究者修改了默认实现的FPFH描述符,将8个药典特征在一个点附近的分布编码成8-bin直方图(表2),每个bin对应8个药典特征中的一个。接下来使用最终的41-bin直方图,即c-FPFH来改进BO1腔对RANSAC的初步比对。使用ph4-ext评分函数对BO1腔对的对齐进行评分,在区分相似对和非相似对方面,新型c-FPFH明显优于标准方法(c-PFPH, ROC AUC= 0.93, CI = [0.91;0.94]; FPFH, ROC AUC = 0.87)(图3)。在相同的数据集上,该描述符的性能几乎与使用最先进的Shaper对齐工具获得的性能相似(ROC AUC = 0.92, CI = [0.90;0.93])。
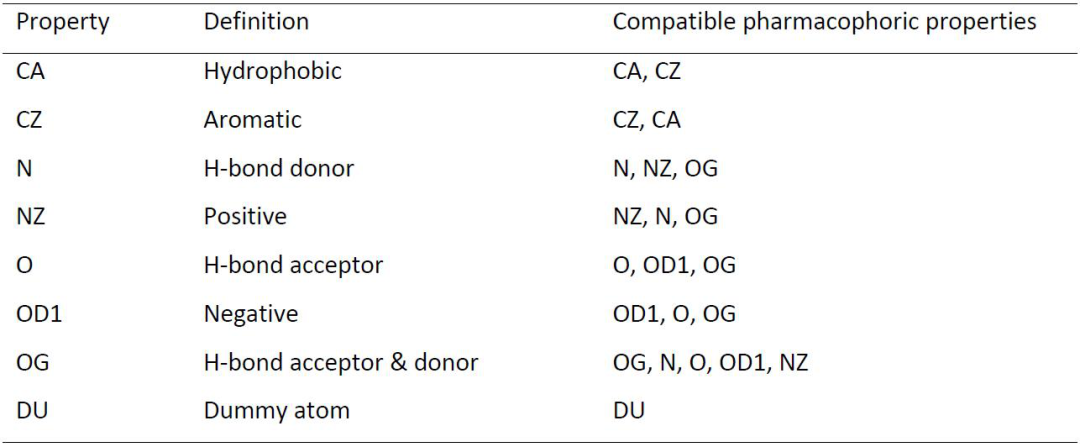
表2 ph4-rules评分函数使用的药典匹配规则。
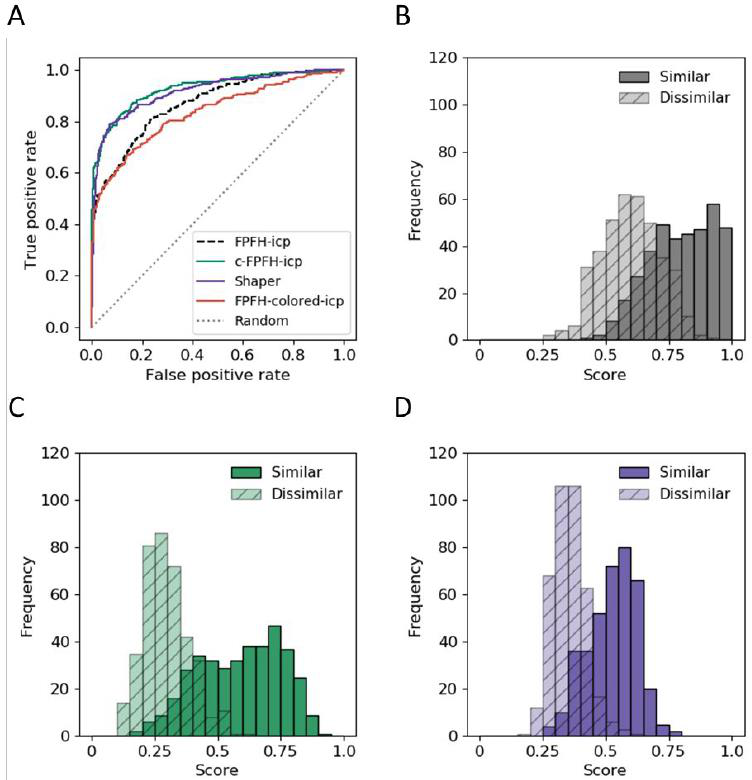
图3 在比较BO1组空腔时的ProCare评分的评价。
c-FPFH描述符辨别能力的提高是由于纠正了之前报告的对齐误差,结果反映在分数上。失调的另一个原因是形状的差异(球状比平面)观察两腔之间。呈现相似性的随机采样点难以捕捉。当然,不能排除错误注释BO1对的可能性,特别是那些预测的不相似。然而,观察到功能不相关的蛋白质结合位点之间的相似性是非常罕见的事件,因此,即使在数据集中存在这种情况,也可以忽略不计。
ProCare评分分布的统计评价。结合c-FPFH描述符进行对齐和ph4-ext进行评分的方法的能力,首先通过使用ph4-ext评分的增量变化(从这里开始计算ProCare评分)区分BO1集合的相似和不同腔区的能力进行评估。在调查数据集的阈值为0.39的情况下,得到最佳判别能力(recall = precision = F-measure = 0.85)(图4A)。为了检查该阈值是否与数据集相关,研究者生成了250万个对齐的背景分布(510个非冗余BO1空腔与4,223个scs -PDB空腔)。根据Kolmogorov-Smirnov检验(D = 0.046, P-value = 0.0292,α=0.02),每100,000个值的100个统计代表性样本,可以拟合到一个广义极值(GEV)分布(图4B)。
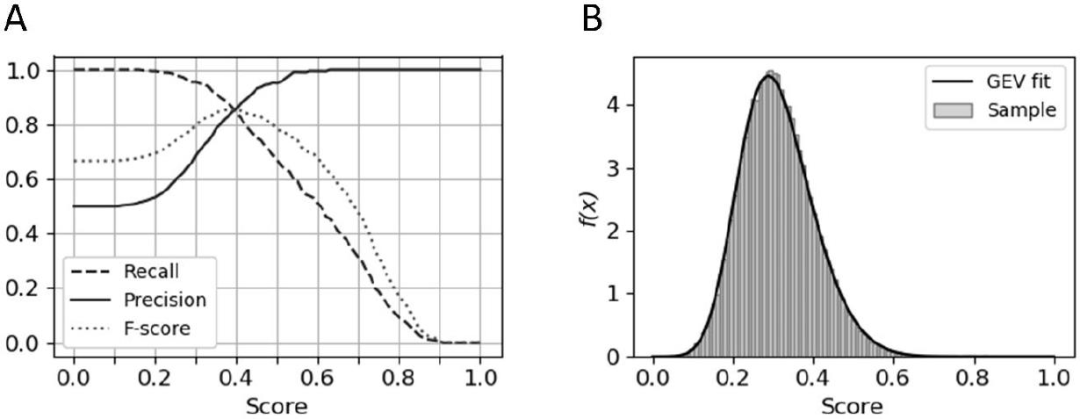
图4 原子坐标变化的统计评价和灵敏度。
从背景分布来看,ProCare评分有统计学意义的阈值为0.47,对应的p值为0.05。在这个阈值下,前一个BO1集合的分类的召回率较低(0.72),但精度高得多(0.95)。从这里开始,ProCare将与上述报告的最佳参数集一起使用,将c-FPFH描述符用于对齐,将ph4-ext用于计分袋对齐。
标杆法与药物化学中最先进的方法。研究者在此选择了一个经典的药物化学方案:两个口袋结合相同的配体(化学类型)或不?为此,研究者重新访问了最近发布的顶点数据集,其中包含6,029个蛋白结构定义的6,598个阳性蛋白对和379个阴性蛋白对。然而,已发布的数据集存在强烈的不平衡(正对>>负对),需要进行一些过滤,才能达到等效的338对阳性和338对阴性。六种公开可用的方法(FuzCav, Kripo, PocketMatch, ProBiS, Shaper, SiteAlign)被不同的组认为是最先进的腔体比较工具,通过简单估计配体结合口袋相似度,与本文提出的方法进行比较,以区分阳性和阴性对(图5)。
总体的趋势是,将物理化学和/或药物学性质映射到结合位点原子上的方法(FuzCav, PocketMatch, SiteAlign, KRIPO)优于两种方法(ProCare, Shaper),它们依赖于将描述符映射到赝配体和原子坐标上。
但是,这些工具都有自身的缺陷所在(而ProCare不存在):
1. 不依赖-比对的方法(FuzCav, PocketMatch)非常快速和准确,但由于没有生成蛋白质覆盖,结果很难解释。从药物化学的角度来看,蛋白质配位的缺失阻止了配体从一个参考口袋转移到另一个参考口袋,从而阻碍了基于结构的靶标-到-先导物的优化。
2. SiteAlign技术虽然非常精确,但速度非常慢(约30秒/对比),并且对较短的蛋白质列表的适用性有限,除非在分布式并行计算环境中执行。ProBiS允许对正负对进行精确分类,但代价是完整性较低(只有64%的对可以处理,图5)。
3. 依赖已知蛋白配体相互作用生成结合位点描述符的KRIPO方法在5%的测试案例中未能产生结果,也不能用于apo蛋白。
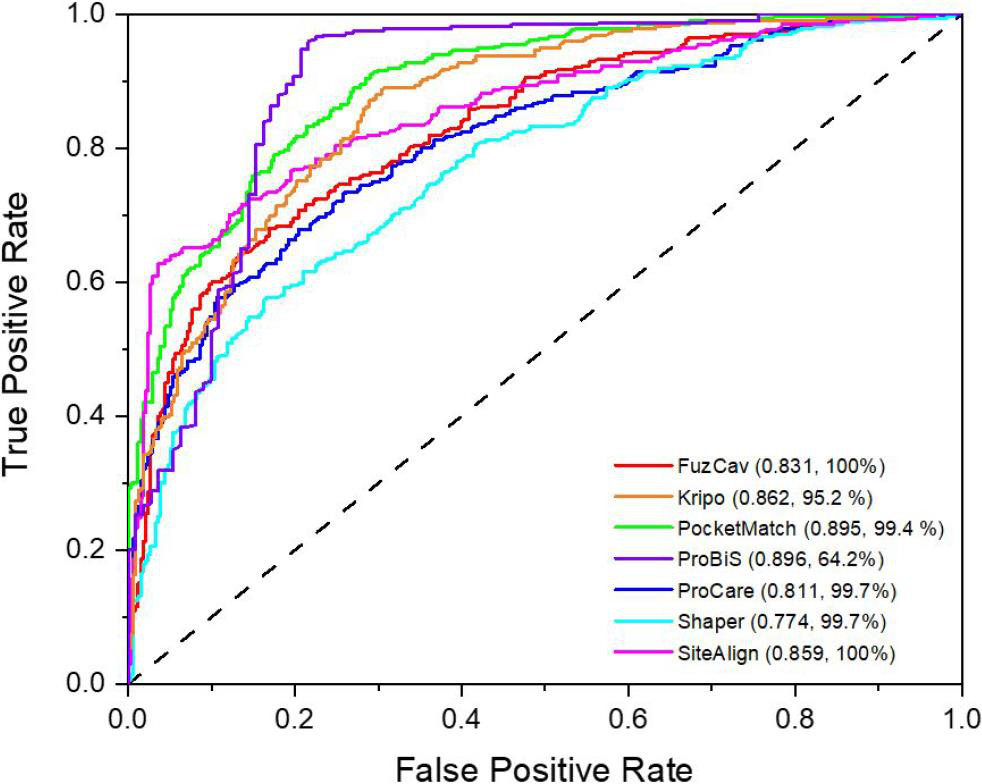
图5 根据六种不同的方法,通过减少口袋相似度来绘制676个蛋白对(顶点集:338个阳性,338个阴性)的受试者工作特征图。每一种方法的ROC曲线下面积和完满度(处理成功对的百分比)在括号中表示。
因此,ProCare是一种广泛适用的、健壮的检测结合位点相似性的方法,因为它是唯一累积速度快(几秒/对比),精度好的方法(ROCAUC = 0.81),可解释性(对齐蛋白,匹配残基之间的距离列表)和大的应用范围(配体结合和游离配体蛋白结构)。
检测片段子口袋和全蛋白空腔之间的相似性。 从以上可知,点云配准可成功地来对齐和比较整个蛋白空腔。那么,它适用于较小的物体(片段-绑定位置)吗?因此,研究者系统地将同一蛋白结合到类药物配体或后期配体的亚结构片段中的Frag-Lig组中的空腔对排列起来。将ProCare变换矩阵应用于相应的蛋白质-片段复合物,并计算两种性质,就可以很容易地推导出全腔对齐的正确子口袋:(i)片段结合蛋白到完整配体结合目标的rmsd;(ii)观察到的全腔与合并片段或参比全类药物配体之间相互作用的相似性。
用相应云中的点数来表示的口袋大小,证实了片段结合的子口袋比相应的全配体结合的整个空腔要小得多。在91%的案例中,通过组合延伸(CE)方法对两个蛋白结构进行结构对齐,当与原始的片段结合蛋白结构相比,在低于2 Å的C-α原子上产生rmsd,说明配体结合后蛋白水平上没有发生重大构象变化(图6A)。在这种情况下,ProCare在提出可靠对齐方面(蛋白质主链原子2 Å的rmsd)明显优于Shaper,在42%的情况下优于基于高斯的Shaper方法的34%(图6A)。对于那些结构对齐良好的口袋,98%的情况下ProCare得分高于之前定义的阈值(0.47分,p-value = 0.05),这表明通过对齐满腔获得的分数可以转化为不同大小口袋的比较。
接下来,研究了ProCare提出的更好的对齐是否与旋转/平移到全空腔后碎片的更好定位相对应。研究者利用基于分子相互作用指纹图谱(IFP)计算的Tanimoto系数,估算了片段子口袋与原拟对齐片段或天然药物样配体之间相互作用的相似性。
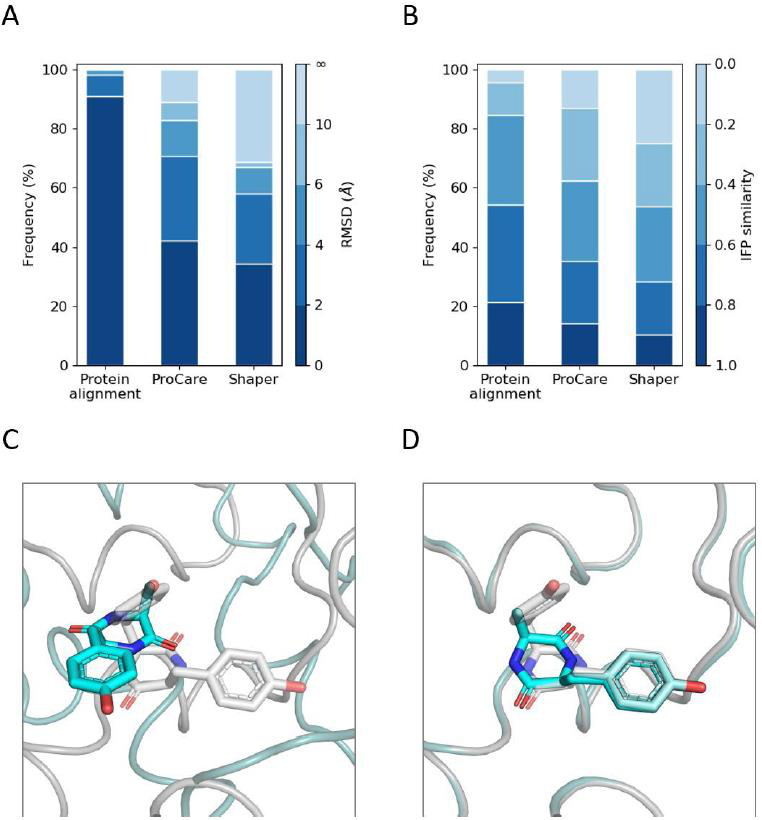
图6 对片段超囊与满腔的ProCare排列的评价。
考虑到IFP相似度高于0.6的保守结合模式,CE结构比对表明,53%的情况下片段结合模式保守在完整配体中(图6B)。在此基础上,ProCare在35%的案例中成功地将碎片准确定位在全口袋中,而Shaper仅在28%的案例中成功定位(图6B),因此证实了ProCare提供的更好的腔体排列也可以转化为更好的碎片姿势。在许多例子中,通过此处描述的点云配准,确实挽救了Shaper的不对称(图6C, D)。
虚拟筛选片段子口袋,以协助基于片段的药物设计:第一个概念证明。接下来,研究者扩展了片段定位的概念,对不相关的蛋白质,从结合位点比对推断。在基于片段的药物设计中,研究者采用了最近首次在蛋白质数据库中公开的蛋白质配体复合物的高分辨率X射线结构,并检查是否筛选一组片段子口袋以获取与新查询空腔的相似性(表3),帮助重构,甚至部分重构被掩盖的查询结合配体。
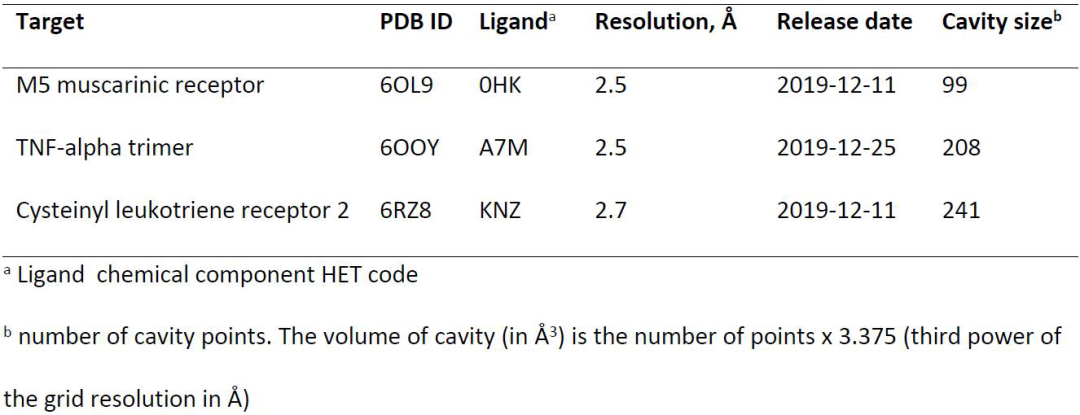
表3 比较最近在PDB中释放的三种蛋白配体复合物的结合位点。
通过分割所有sc-PDB结合配体(sc- PDB片段集),同时保持蛋白质结合的三维坐标,获得了33,953个片段子口袋。然后对片段库收集进行筛选,确定了与三个新空腔的ProCare相似性。点云配准后,使用最优变换矩阵将对应的片段合并到查询腔的坐标框架中,并根据两项条件进行滤波:(i)遵守片段三规则;(ii) ProCare 评分 > 0.47。将α选择的片段与与目标查询共结晶的掩蔽配体进行比较时,考虑口袋相似度和交互指纹相似度,将剩余片段命中次数按综合评分(FragScore)进行排序。
第一个查询是作为ProCare校准协议和随后的评分函数的质量控制。因此,三个基于硫托溴铵的片段被排在前33位片段中,相对于真正的M5-束缚的硫托溴铵位姿,它们的形态很好(图7A,表4)。有趣的是,来自配体与不相关蛋白结合的高级别片段(如血淋巴保幼激素结合蛋白,PDB ID: 3AOS,配体HET: JH2;组蛋白去乙酰样酰胺水解酶,PDB ID: 1ZZ1,配体HET: SHH;图7B,表4)很好地与M5-束缚的噻托溴铵重叠,并为片段生长和/或连接提供了合适的起始点。第二个查询腔(681 Å3)位于不对称肿瘤坏死α-因子(TNF-α)三聚体的界面。然而,从不相关的蛋白质中选择的几个sc-PDB片段(如4KZ0_1UJ, 3R04_UNQ)出现在顶级ProCare评分者中,并且是TNF-α抑制剂的苯并咪唑部分的真实生物电子等排体(图7C-D,表4)。所选片段的ProCare姿势很好地与真正的配体重叠,并概括了芳香族相互作用所展示的双环苯并咪唑环和一个氢键到TNF-α腔的Tyr151侧链。同样,真正的TNF-α抑制剂的双取代芳香族取代基也被得分最高的芳香族片段模仿(3N6U_NSU,图7E,表4)。
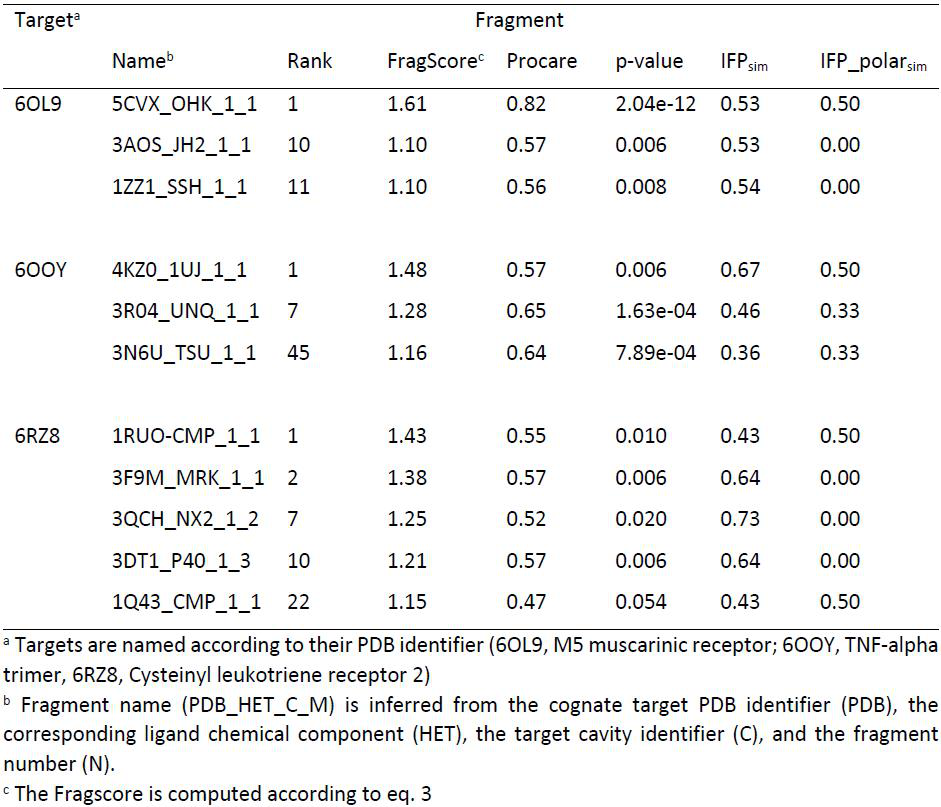
表4 为三种新型腔体选择最顶端的片段。
展望与结论
研究者提出了一种新的叠合和比较蛋白质空腔的计算方法ProCare,空腔被表示为3D点云,由模仿理想配体的药效学特性进行注释,并通过点云配准进行排列。重要的是,ProCare利用了一种新的点特征直方图来编码腔微环境,从而有利于共享相似几何和物理化学性质的子口袋的叠加。ProCare仍然可以在速度和完整性方面进行优化。两两相似度搜索可以在几秒钟内完成,但通过优化最近邻搜索和排除初始RANSAC对齐过程中的不相关点,可以显著降低CPU成本。
参考文献:EGUIDA, M., & Rognan, D. (2020). A computer vision approach to align and compare protein cavities: Application to fragment-based drug design. J. Med. Chem. 2020, 63, 13, 7127–7142. DOI:10.1021/acs.jmedchem.0c00422