
引言
卡纳万病(CD)是一种罕见的疾病,这种疾病是由aspA基因突变引起的,该基因编码了天冬氨酸酰基酶(ASPA)。这些突变导致氨基酸取代,产生突变酶异构体的表达,从而导致了酶的不稳定或催化活性降低,导致关键脑代谢物N -乙酰- l-天门冬氨酸(NAA)的积累,并导致了少突胶质细胞中醋酸盐的生产不足,从而会导致脂肪酸生物合成的减少和神经元的脱髓鞘。截至目前,由于缺乏有效的治疗方法,CD仍然是一个致命并且无法治愈的疾病。
先睹为快
作者信息
美国加州旧金山Atomwise公司、美国俄亥俄州托莱多大学化学与生物化学系
Adrian Stecula、Ronald E. Viola
靶点
天冬氨酸N-乙酰转移酶ANAT
计算方法
同源模建,深度卷积神经网络AtomNet
计算流程
研究者基于低蛋白质序列同源性“模糊区域”的模板结构生成了同源模建模型,随后筛选了商业化合物库Mcule(约1000万化合物)。去除分子量在100-800以外的,手性超过7个,以及可旋转键大于16的分子,然后利用RDKit中的PAINS进行过滤,剩下7 177 223分子经过AtomNet(深度卷积神经网络)打分,打分前20000的分子,选择分子量大于200,并且不含对特定基团的化合物,随后进行结构聚类,选择743个代表性化合物,随后在MolSoft ICM软件中,按照molecular weight<550 Da, logP<7, PAINS score<0.6, toxicology score<2.5, “bad groups”= none进行过滤,选择打分前111个化合物进行购买,其中60个化合物在DMSO中具有较好的溶解性,选择这60个化合物进行实验验证高置信度预测的结合物的体外测试,从中发现了靶向ANAT的类药性小分子抑制剂。
过去几十年里,绝大多数针对CD的治疗方法的研究都集中在ASPA上。ASPA是一种有缺陷的酶,专门负责将NAA代谢成大脑中的天冬氨酸和醋酸盐。最近的研究提供了令人信服的证据,证明导致CD症状的主要原因是脑内NAA水平升高,而不是无法产生下游代谢物。脑内NAA的合成是由nat8l基因编码的天冬氨酸N-乙酰转移酶(ANAT)催化的(图1A)。敲除nat8l基因可以消除作为CD标志的大脑缺陷。免疫组织学和TEM成像显示,没有证据表明CD中存在广泛的空泡化和脱髓鞘,NAA水平的降低减少了大脑皮层和小脑区域神经元的损失。尽管这些动物将NAA代谢成天冬氨酸和乙酸盐的能力减弱,但发育缺陷的消除仍然发生,这是代谢的结果,也是大多数CD治疗研究的焦点。通过降低NAA水平,不仅可以消除这些发育缺陷,而且行为和表现评估表明,与受损的卡纳万小鼠模型相比,这些缺陷有显著改善。
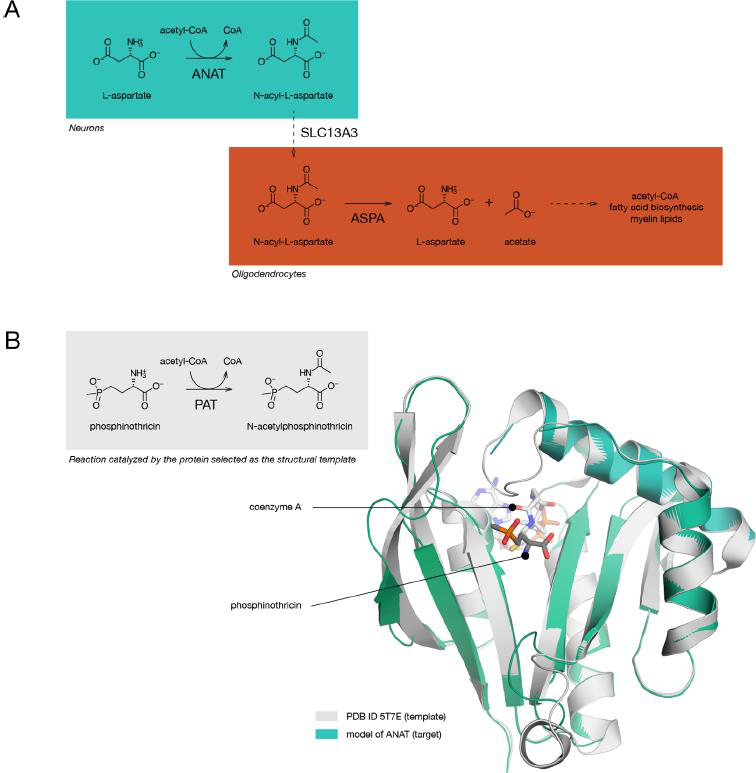
图1脑内NAA的代谢及同源模建构建的人ANAT模型
图片来源JMC
当前,阻碍进一步研究CD患者NAA水平下降的影响的一个关键问题,是无法获得ANAT和缺少工具化合物来探究这一途径。与此同时,目前市场上还没有FDA批准的ANAT抑制剂。目前已知的抑制剂是通过片段库筛选确定的,随后迭代优化产生该酶的第一个有效抑制剂。但由于细胞通透性和潜在毒性,临床上并不受欢迎。在这里,研究者描述了一个新的方法可用于ANAT的靶向识别。研究者基于低于蛋白质序列同源性“模糊区域”的模板结构生成了一个比较结构模型,随后使用AtomNet(深度卷积神经网络模型)筛选了包含近1000万化合物的现成商业化合物库。经过体外测试发现了靶向ANAT的一类小分子抑制剂。
AtomNet训练
AtomNet是第一个用于基于结构的药物发现的深度神经网络。使用几百万个小分子的生物活性值(例如Ki或IC50)和几千个不同家族的蛋白质结构,以此训练形成一个单一的全局AtomNet模型,用来预测结合亲和力。
训练过程如下:
1. 使用基于初始起始点的泛洪算法在给定的蛋白质结构上定义结合位点。可以通过多种方法来确定,包括RCSB PDB数据库中标注的结合配体,诱变研究中发现的关键残基,或文献中报道的催化基序的鉴定。
2. 将共复合物的坐标转换为以结合点的质心为原点的三维笛卡尔坐标系。然后,通过围绕结合位点的质心随机旋转和翻译蛋白质来进行数据增强。
3. 随后,对于一个给定的配体,研究者在结合位点腔内对多个姿态进行采样。每一个姿态都代表一个假定的协复合体,因此,与以往基于结构的预测方法(如对接)不同,该方法不需要实验协复合体来进行训练或预测。
4. 然后将生成的每个共复合物栅格化为固定大小的规则三维网格,其中每个网格点上的值表示每个网格点上存在的结构特征,不同网格点表示不同的原子类型(或更复杂的蛋白质配体描述符,如SPLIF、SIFt以及APIF等)。这些网格作为卷积神经网络的输入,定义了网络的接受域。研究者采用了一种输入层间距为1 Å的30 × 30 × 30网格的网络架构,随后是六个依次是32 × 33, 64 × 33, 64 × 33, 64 × 33, 64 × 33, 32 × 23((滤芯数量×滤芯尺寸)的卷积层,最后是一个全链接层以及ReLU激活单位。每个姿态的得分会通过一个加权的波尔兹曼平均值进行组合,这个加权平均值会根据他们的预测得分对姿态进行加权。然后将这些分数与实验测量的蛋白质和配体对的活性等级进行比较,并用二元交叉熵损失函数调整神经网络的权重以减少预测和实验测量的亲和力之间的误差。使用ADAM自适应学习方法、反向传播算法和每梯度步长64个生物活性目标化合物对的小批量进行训练。
预测遵循一个类似的过程。研究者在靶蛋白上选择一个正位或变位结合位点。接下来,对于给定的感兴趣的分子筛选库中的每个分子,生成一组结合位点内的位姿。每一个都由训练过的模型打分,分子列表也根据它们的分数进行排序。然后,根据标准化的过滤协议、供应商的可用性和价格,研究者购买列表顶端的一组分子进行了实验测试。该操作不会通过对化合物的视觉检查而引入人工偏差。
同源模建
在项目开始的时候,还没有公开的人类ANAT蛋白的结构。研究者使用SWISS-MODEL对模板结构的搜索表明,序列识别范围在9% (PDB 编码4RI1)到25% (PDB 编码3PP9)之间存在许多同源结构。经过大量的分析研究,最终,研究者选择PDB code 5T7E作为比较结构建模的模板,原因是结构中存在底物以及两种蛋白的催化活性相似(图1B)。人类天冬氨酸N-乙酰转移酶依赖乙酰辅酶A作为乙酰基供体来执行其催化功能。该酶的催化区域预测为假设常见的Gcn5相关N-乙酰转移酶(GNAT)折叠。利用PROMALS3D构建目标模板序列比对。对比结构模型在Molsoft ICM 3.8中使用完整的模型构建器模块和完整的优化例程中建立而成,保留辅酶A和L-草丁膦分子作为刚体。人工检查模型,确保侧链原子没有缺失时,添加氢,将辅酶A分子替换为PDB code 5T7D的乙酰辅酶A。以L-草丁膦分子为起始点,采用泛洪算法确定筛选位点。
虚拟筛选过程
从一家化学品供应商(Mcule, https:// mcule.com)获得约1000万商用化合物。使用RDKit,去除盐,规范化结构,去重。研究者还过滤了100和800 Da范围之外的分子,包括那些包含超过7个手性中心的分子,以及大于16个可旋转键的原子。最后,利用RDKit中实现的PAINs过滤器过滤所有剩余的化合物。剩下的7 177 223个分子由AtomNet在Amazon Web Service((https://aws.amazon.com)基础设施上的一个由GPU和CPU组成的弹性混合集群上对目标进行打分。得分最高的且分子量 > 200 Da的2万个化合物随后被进一步过滤,使用一套专有的SMARTS模式进行过滤,其中包含类药物化合物中通常认为不需要的部分(如冠醚、碳二亚胺)。剩余的化合物使用ECFP4指纹Tanimoto系数相似度截止值为0.3的Butina聚类算法进行多样性聚类。最后一组743个集群被导入并在MolSoft ICM 3.8中采用以下标准:分子量小于550 Da, log P < 7、PAINS评分< 0.6,毒理学评分 < 2.5,不良基团=无。最后打分前111个分子作为代表性分子将用来购买。其中60个被发现可溶于DMSO,并通过了内部质量控制程序,这些化合物可供实验验证。
实验验证
实验分析基于Viola实验室开发的自定义协议。每一种购买的化合物都在1 mM的初始筛选中进行了测试。其中7种化合物显示出了显著的酶抑制作用,而60种化合物中约有一半在如此高浓度的条件下,即便在DMSO存在的情况下,溶解性仍然有限。第二次筛选的化合物是在50 μM浓度下进行的。实验显示,没有一种化合物显示出明显的沉淀。在这个较低的浓度下,7个初始靶向物中的5个仍然被观察到对酶有抑制作用。随后在剂量反应方案中对这5种化合物进行了评估,每个化合物都在亚微摩尔范围内显示出Ki值。
为了研究化合物的溶解度和聚集性,在低浓度洗涤剂的存在下重复了剂量反应实验。当在该洗涤剂存在的情况下进行检测时,每种化合物测定的抑制作用都有所改善,与不存在该洗涤剂时测定的值相比,Ki值提高了2- 4倍(表1)。
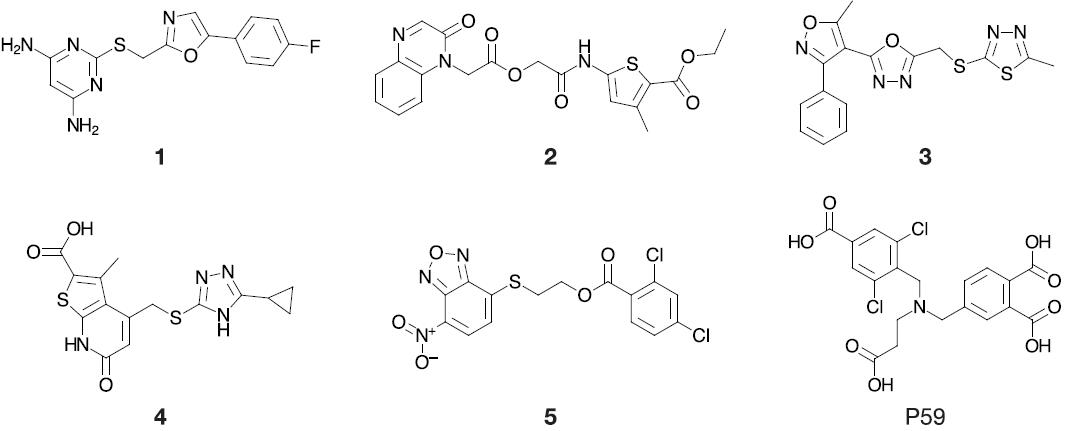
图2 化合物的结构(1-5)经AtomNet鉴定,并在体外验证为ANAT抑制剂
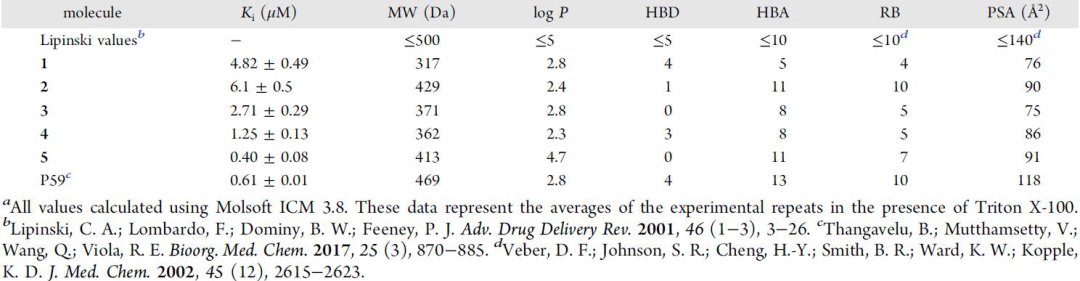
表1 靶向分子的分子性质
展望与结论
在这项研究中,研究者筛选发现了5个亚微摩尔级活性的天冬氨酸N-乙酰转移酶抑制剂。这些化合物有潜力作为未来治疗发现项目的起点。同时,研究者展示了AtomNet在具有挑战性的约束条件下支持早期药物发现的能力。首先,研究者证明了即使在目标数据稀缺或完全不可用的情况下,AtomNet仍然能够发现新的活性骨架。其次,研究者证明了即使没有三维晶体结构或高序列同源模板的可用性,发现类药分子也是可能的。最后,研究者证明了在极其有限的资源下,研究新药物也是可能的,机器学习可以帮助人类降低难度。在这项研究中,研究者从1000万化合物中只购买并测试了60个化合物,深度学习的引入大大降低了化合物筛选的难度。
参考文献
Stecula, A., Hussain, M. S., & Viola, R. E. (2020). Discovery of Novel Inhibitors of a Critical Brain Enzyme Using a Homology Model and a Deep Convolutional Neural Network. Journal of Medicinal Chemistry. DOI: 10.1021/acs.jmedchem.0c00473