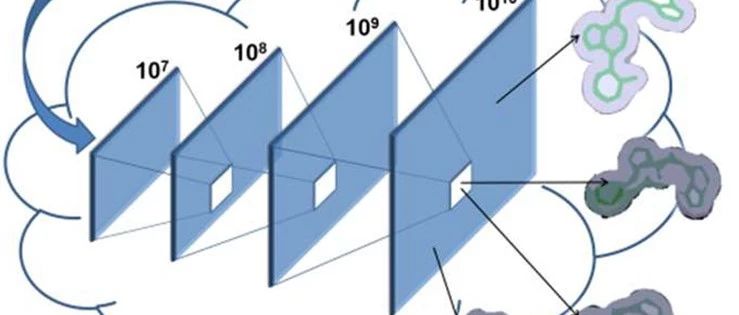
虚拟筛选

虚拟筛选是利用计算机模拟药物筛选过程,对化合物的活性做出预测,可从大型化合物库中迅速筛选出有潜在活性的候选药物。目前用到的虚拟筛选方法包括基于结构的药物设计(SBDD)、二维指纹相似性、三维形状的相似性,其在准确性、速度和新颖性方面都有优缺点(如图1)。
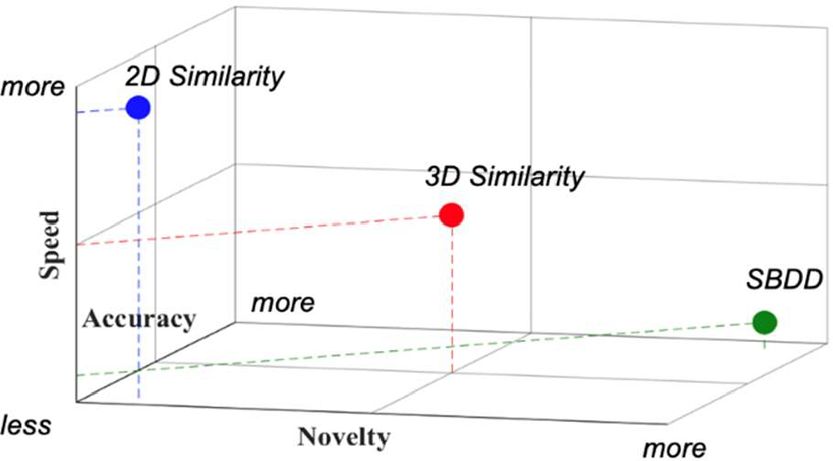
图1.二维相似性(蓝色)、三维相似性(红色)和基于结构的药物设计(绿色)的虚拟筛选方法在准确性、速度和新颖性方面的定性比较
图片来源JCIM
三维形状相似性搜索的虚拟筛选
本文集中讨论了三维形状相似性搜索的虚拟筛选,以及如何将其扩展到非常大的化学空间,进一步研究了需要列举和搜索多少化合物,以最大限度地从给定化学空间中检索新的化合物,达到高效率高质量的目的。ROCS是一款基于三维形状相似性的虚拟筛选工具,通过形状比较快速地识别潜在的活性化合物,其中,FastROCS是目前基于3D形状的最快方法之一。然而,生成和搜索超过几百万个分子的三维构象数据库可能需要大量的硬件资源,使得本地计算机资源紧张。在本文,作者是在OpenEye云平台Orion中的FastROCS,实现了10^5-10^10数量级虚拟分子库的生成和搜索,并讨论了五个数量级的分子库虚拟筛选的时间、费用、命中率和相似性得分的差异。虚拟分子是基于4100多个已建立的合成路线生成的,如图2所示,通过三个分子片段组成一个全新的虚拟分子,根据给定的化学路线大部分都能被合成出来。
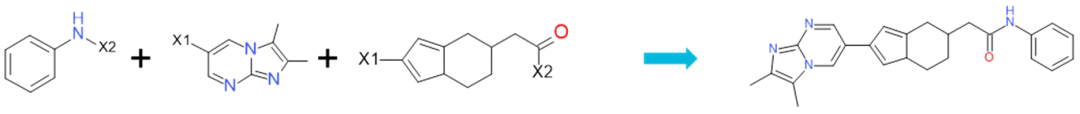
图2. 虚拟分子产生的示例图
图片来源JCIM
结果
为了进一步评估FastROCS虚拟筛选的性能,作者生成了分子量为51M、172M、1330M和12700M四个数据库。图3给出了生成数据库的时间和费用,以及基于云平台的FastROCS搜索这些数据库所需的时间。计算费用与分子库数量有明显的线性关系,因此可以根据计算的数据库大小预估成本。生成数据库所需的时间取决于CPU的数量,使用更多的CPU可以很容易地降低所需的时间。
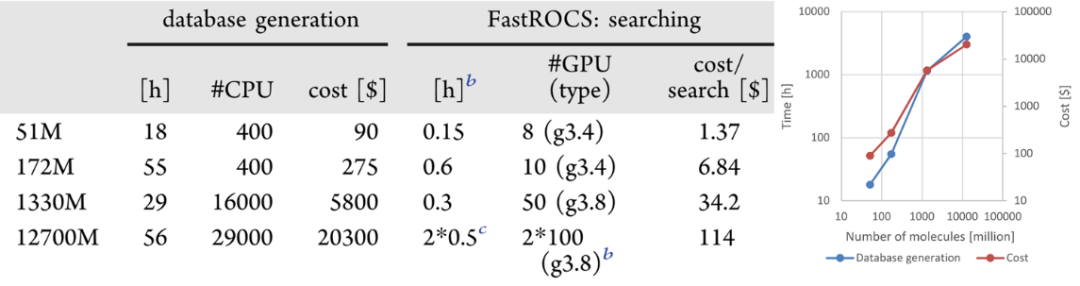
图3. 数据库生成和搜索所需的时间和费用
图片来源JCIM
作者用了十个不同类型的提问式 (query)(图4)查询上述数据库,并统计了排名top200000的分子的命中率,TanimotoCombo相似性量度(Tc),片段的多样性(图5)。如预期的那样,基于查询式得到的分子命中率与分子库大小成线性关系,另外,有趣的是,Tc得分值也随着搜索空间增加而增加。最重要的发现是,用更大的搜索空间检索会增加命中化合物的结构新颖性,并且在10^9-10^10尺度下,曲线变平,即新型骨架的数量不再线性增加。这表明,在10^10尺度达到这个特定化学空间的多样性极限。
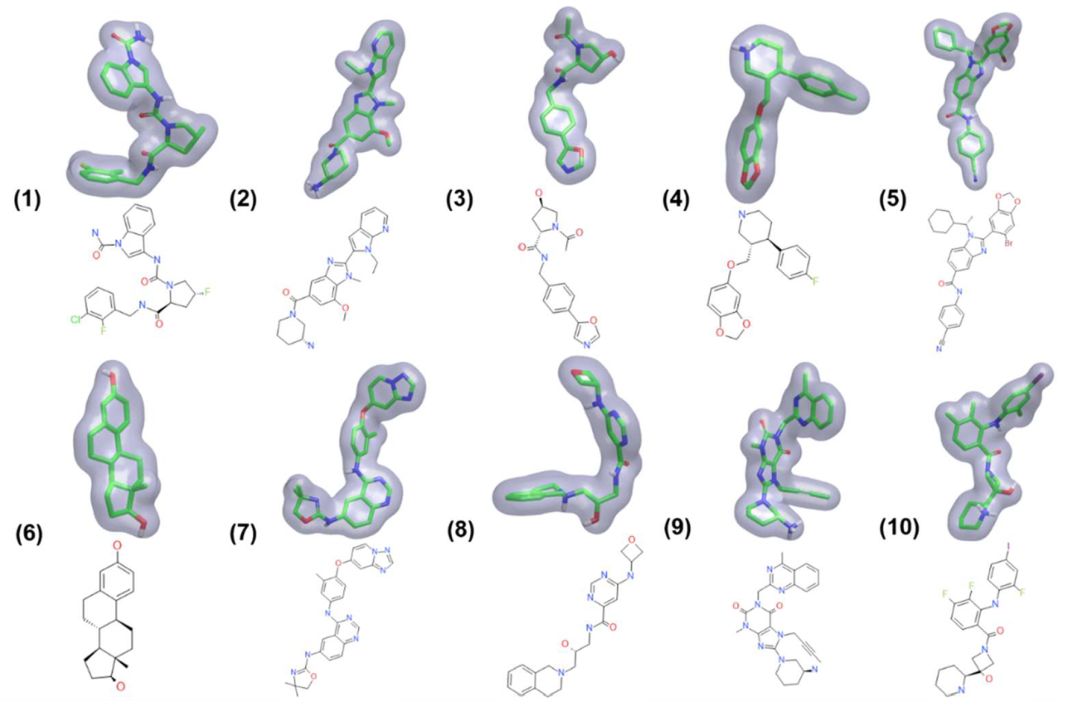
图4. FastROCS搜索中使用的十个查询式的3D形状和化学结构
图片来源JCIM
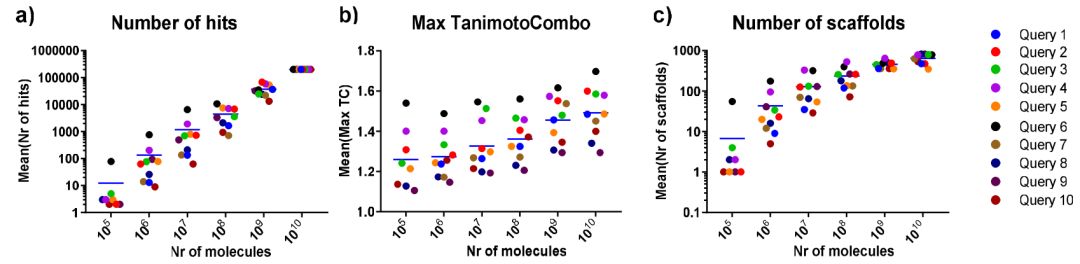
图5. 10个定义的查询式搜索数据库的结果统计
图片来源JCIM
总结
作者通过研究发现,10^10个化合物库足够最大限度地检索新的化合物,不过未来,有待通过优化分子库达到更高的数量级。如果生成一个10^10分子构象集,时间为2-3天,成本约为20000美元。一旦生成,此资源可用于无限数量的搜索,如果用FastROCS对如此大量的分子进行搜索是可以在几分钟内实现的,每个查询大约需要100美元。
参考文献:
Grebner C, Malmerberg E, Shewmaker A, et al. Virtual screening in the cloud: How big is big enough?[J]. Journal of chemical information and modeling, 2019.