
引言
外排转运蛋白P-糖蛋白(P-gp)负责从细胞中转运包括药物分子在内的多种分子。因此,P-gp介导的外排转运限制了药物的生物利用度。为了在药物发现过程的早期识别潜在的P-gp底物,已经基于结构和物理化学描述符开发了计算机模型。本文中,作者研究了分子动力学指纹(MDFP)作为正交描述符用于训练机器学习(ML)模型来将小分子分类为P-gp的底物和非底物。MDFP对来自不同环境(水,膜或蛋白质口袋)中分子的短时间MD模拟的信息进行编码。在内部数据集(3930种化合物)和来自ChEMBL的公开数据集(1114种化合物)上对MDFP的性能作了评估,并且与常用的2D分子描述符(包括基于结构和基于属性的描述符)进行了比较。作者发现,所有测试的分类器均能很好地插值,从而在化学多样化的子集上实现了较高精度。但是,通过使用外部验证和前瞻性分析对模型进行挑战,作者发现只有在MDFP或基于属性的描述符上训练的基于树的ML模型才能较好地将化学空间推广到训练集未涵盖的区域。
背景介绍
P-糖蛋白(P-gp)通过抵消通过细胞膜的被动扩散,在大脑可利用性和耐药性中起着关键作用。其在血液组织屏障尤其是血脑屏障中高水平表达,阻止异种生物以及潜在的CNS药物进入中枢神经系统(CNS)。此外,已经发现P-gp在抗性肿瘤细胞中的过表达与化学治疗药物浓度降低相关。由于P-gp影响药物的药代动力学,因此新型候选药物的外排转运研究已成为药物发现项目早期的重要步骤。目前已经开发了广泛的计算模型,包括基于结构特征或理化性质的简单规则,基于结构的对接方法以及定量的结构-活性关系模型。已经知道药物成为P-gp底物的可能性与其物理化学性质以及其他参数(如氟原子数)相关。许多基于多参数的计分方法,尤其是针对中枢神经系统药物的计分方法,以帮助合理设计大脑可利用性和细胞可渗透性药物。尽管进行了所有这些努力,但由于准确的前瞻性预测仍具有挑战性,因此非常需要用于P-gp底物的新颖预测方法。
机器学习(ML)方法到目前为止显示出区分P-gp的底物和非底物的最高预测能力。但是,大多数已发布的ML模型都是在公共数据集上进行训练的,尽管对研究人员来说非常有价值,但通常会受限于它们的大小(通常少于1000种化合物)和数据的异质性。大型且一致的数据集的可用性对于测试ML方法的真正潜力至关重要。
分子动力学指纹(MDFP)最近已被开发为小型有机化合物的替代分子描述符。MDFP在短时间分子动力学(MD)模拟中描述了这些信息,因此可以通过ML方法加以利用(图1)。在开源MDFPtools软件包(https://github.com/rinikerlab/mdfptools/)中实现了从SMILES开始生成MDFP的自动化工作流程。结果表明,在MDFP上训练的ML模型与最新的计算方法(即自由能扰动或COSMO-RS)具有相似的性能,可以预测水合自由能和分配系数。此外,该方法是提交给SAMPL6挑战以预测辛醇-水分配系数的前十名之一。
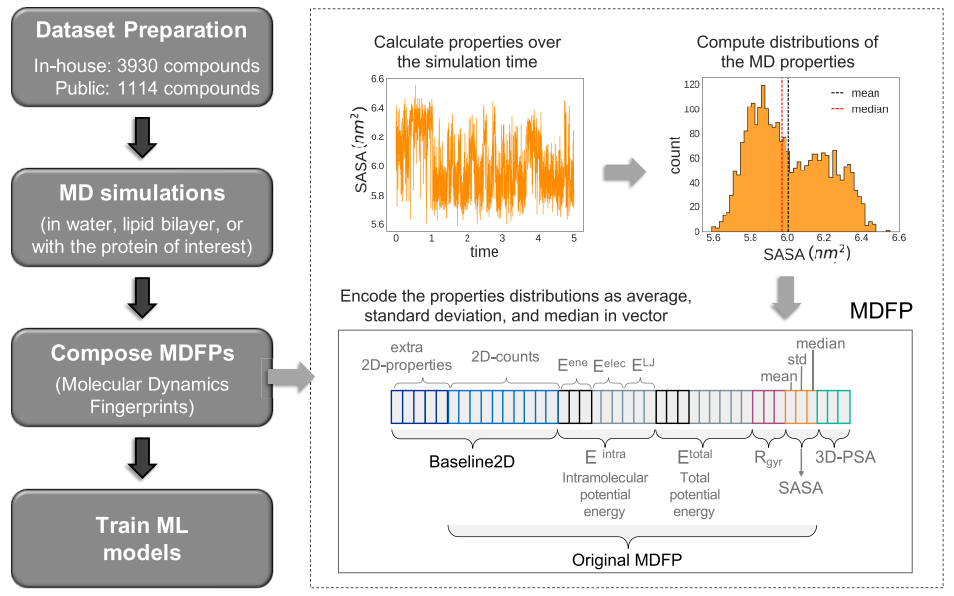
图1 使用MDFPs预测P-gp底物和非底物的计算流程示意图。
图片源自JCIM.
方法介绍
1. 数据集准备:1)内部数据集。对AbbVie数据集(包括9924种8777种化合物的流出比测量值)进行整理和分类(外排比ER≥5的化合物被分类为底物,而外排比ER≤1的化合物被分类为非底物)以及去除某些容易富集(如含硫化合物)而带来潜在的偏向的化合物之后,最终的数据集由3930种化合物组成,其中包含1031种底物和2899种非底物。2)公开数据集。从ChEMBL数据库收集了1114种具有720种底物和394种非底物的P-gp化合物的数据集。3)化学空间分析。使用t-SNE方法在两个维度上映射了两个数据集所覆盖的化学空间。使用ECFP4 Tanimoto系数评估每对化合物之间的距离。
2. MD模拟。对于每种化合物,使用RDKit中的ETKDG构形生成器生成三维(3D)坐标,并使用RDKit中的MMFF力场将其能量最小化。使用ChemAxon插件确定质子化状态和互变异构形式。使用GAFF和AM1-BCC部分电荷对化合物进行参数化。
3. MDFPs的构建。根据化合物在水、脂质双层和P-gp结合口袋中的MD模拟,可以构建不同的MDFPs。从GROMACS轨迹构建MDFPs的功能已包含在MDFPtools软件包(https://github.com/rinikerlab/mdfptools/)中。此外,可以在GitHub(https://github.com/rinikerlab/mdfptools/tree/gmx/tutorials_mdfp)上获得用户教程,以指导用户完成MD模拟的设置、MDFPs的构建以及ML模型的训练。对于膜-溶质MDFP体系,构建了三个MDFP变体,它们包含从POPC双层中的化合物的MD模拟得出的特征(图2):MDFP_POPC,MDFP_POPC *和MDFP_WAT_POPC *。MDFP_POPC描述符由分子内和总能量项、SASA和Rgyr等特征组成;MDFP_POPC *描述符将MDFP_POPC与其他基于MD的特性相结合,这些特性描述了化合物在脂质环境中的特征行为;MDFP_WAT_POPC *是MDFP_POPC *和MDFP的组合(来自水中的MD模拟)。而对于MDFP_PL,除了上述特征外,还计算了溶质与起始构象的RMSD。可以在(https://github.com/rinikerlab/mdfptools/tree/gmx/tutorials_mdfp)上获得蛋白质-配体MDFP的构建教程。
4. 2D分子描述符。使用RDKit基于化合物的2D结构生成所有指纹。包括Baseline2D,PropertyFP,ECFP4和PathFP。
5. ML模型。结合所描述的指纹测试了四个不同的分类器:随机森林(RF),梯度树增强(GTB),支持向量机(SVM)和meta-learner 分类器。
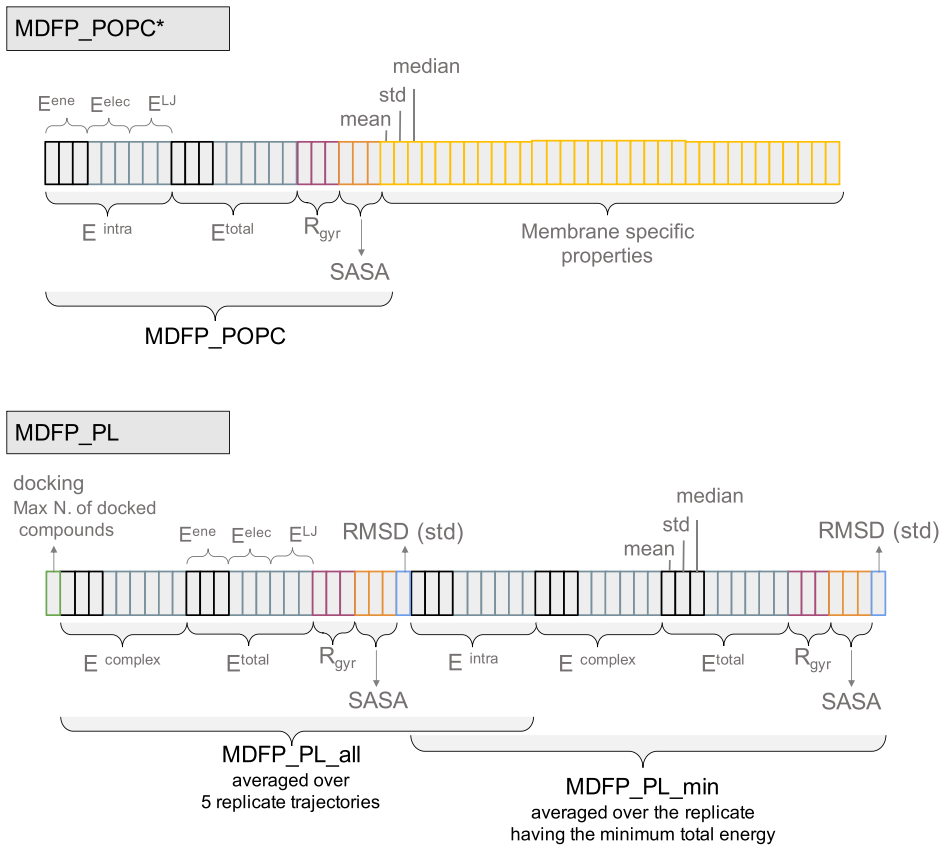
图2 膜-溶质MDFP(MDFP_POPC *)和蛋白-配体MDFP(MDFP_PL)的构建示意图。
图片源自JCIM.
结果1.内部和公开数据集的表征
为了可视化对应于这两个数据集的化学空间,使用ECFP4指纹进行了t-SNE分析。图3A显示两个数据集覆盖了化学空间的不同区域。一些密集区域仅由底物或非底物构成,而其他区域则包含这两类化合物(图3B)。公开数据集主要填充t-SNE图的中心区域,并且没有呈现大分子簇(图3A)。因此,可能很难基于ECFP4指纹将公共数据集的化合物分为底物和非底物。相反,由于内部数据集的化合物在这两个类别之间显示出较少的重叠,因此使用ECFP4描述符可以期望ML模型获得良好的性能。
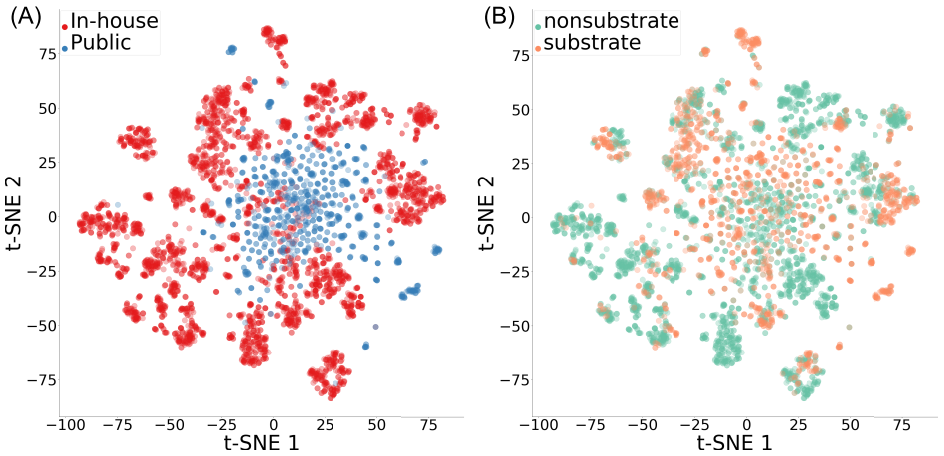
图3 内部和公共数据集覆盖的化学空间比较。
图片源自JCIM.
为了评估底物和非底物分布之间的差异是否具有统计学显著性,我们进行了Welch t检验,我们发现在类别之间显示出更大差异的特征是描述化合物的极性/疏水性、氢键能力和柔性的特征。其中包括2D计数,例如可旋转键的数目以及氢键供体和受体的数目,还包括从水中的MD模拟得出的属性,例如3D-PSA、SASA、分子间LJ势能以及各能量项的标准偏差。不管是内部数据集还是公共数据集,底物似乎比非底物分子量更大、极性更大且更具柔性。
结果2. 评估分类器的预测性能
我们评估了四种不同的ML方法(RF,GTB,SVM和meta-learner)与六种不同的指纹(包括基于属性的指纹、基于MD的指纹和基于结构的指纹)的性能,用于对P-gp的底物和非底物进行分类。内部数据集和公共数据集的不同训练-测试拆分用于评估分类器的准确性、可概括性和预期性能。图4显示了内部数据集的不同模型之间的遗传算法比较。所有分类器均具有较高的预测能力,GA率介于0.78±0.02和0.86±0.02之间(表1)。尽管RF和GTB与PropertyFP或MDFP结合使用可获得更高的精度,但SVM在二进制结构指纹ECFP4和PathFP方面表现最佳。实际上,表现出最好分类结果的模型是SVM/ECFP4_MDFP,其准确度= 0.85±0.01,MCC = 0.66±0.02和AUC = 0.92±0.01。
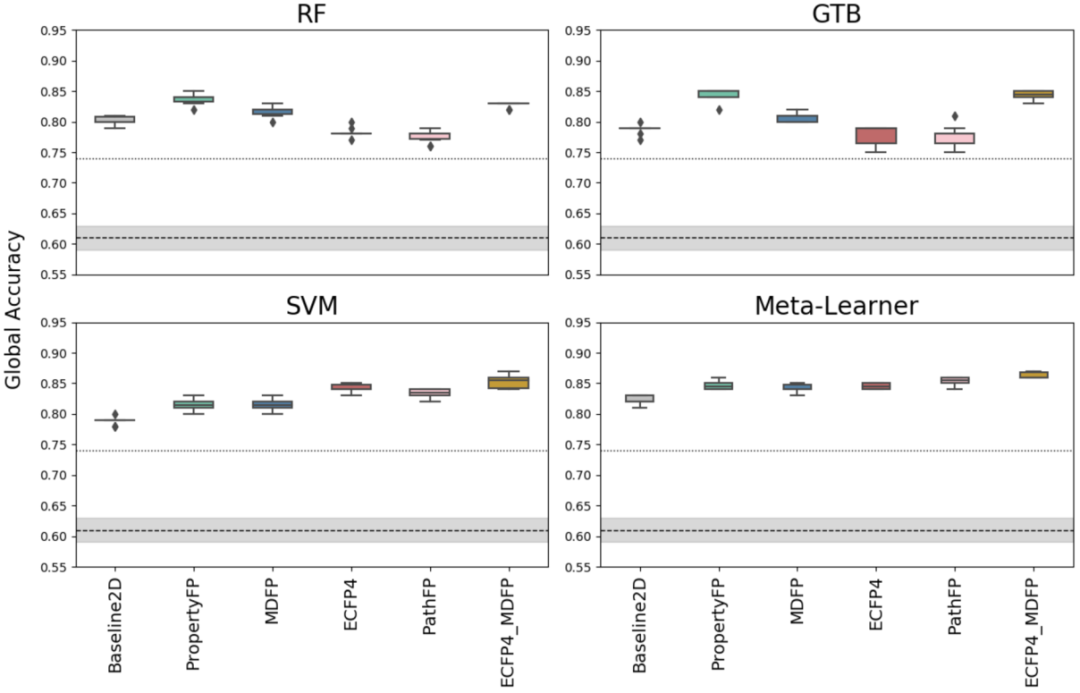
图4 在不同的ML模型和描述符之间针对内部数据集的分层化学多样性拆分而获得的GA的比较。
图片源自JCIM.
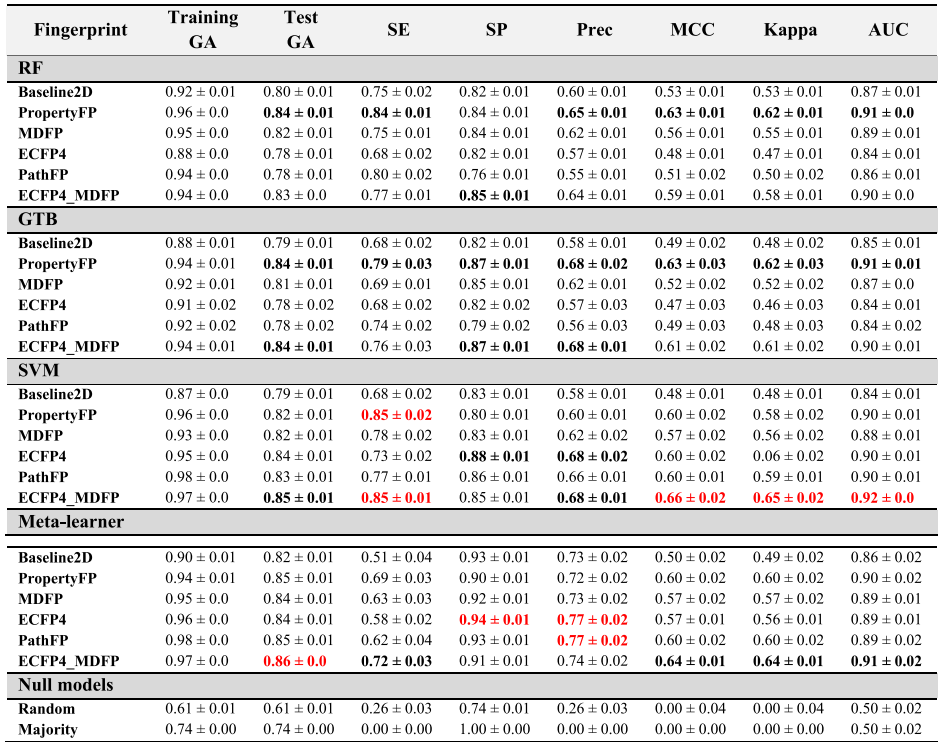
表1. 内部数据集分层化学多样性拆分上不同分类器之间的性能比较。
图片源自JCIM.
结果3. 膜-溶质MDFPs和蛋白-配体MDFPs的性能评估
MDFPs方法的主要优点之一是多功能性,即可以根据要预测的属性或生物学过程对其进行修改和扩展。由于分子模拟的计算成本较高,因此仅针对公开数据集(1140种化合物)以及内部数据集中的1000种化合物的平衡子集生成了膜-溶质和蛋白质-溶质MDFPs。通过使用RDKit中的MaxMin算法来选择化合物,可以最大限度地提高内部子集的化学多样性。膜-溶质和蛋白质-配体MDFPs的预测能力在分层化学多样性拆分上进行了测试。对于公共数据集,采用的决策阈值为0.6。表2列出了RF获得的结果。对于这两个数据集,由MDFP_POPC*和MDFP_PL描述符实现的性能都不错,但并不比上面讨论的水MDFPs更好。实际上,与仅包含15个2D计数和属性的Baseline2D相比,MDFP_POPC *和MDFP_PL的性能明显优于空模型,但给出的结果相似(甚至稍差)。此外,MDFP_POPC和MDFP_POPC *之间的比较表明,增加表征膜中溶质行为的特性不会导致性能的提高,且混合MDFP_WAT_POPC*和MDFP_WAT_PL描述符的性能分别优于单个MDFP_POPC*和MDFP_PL组分。
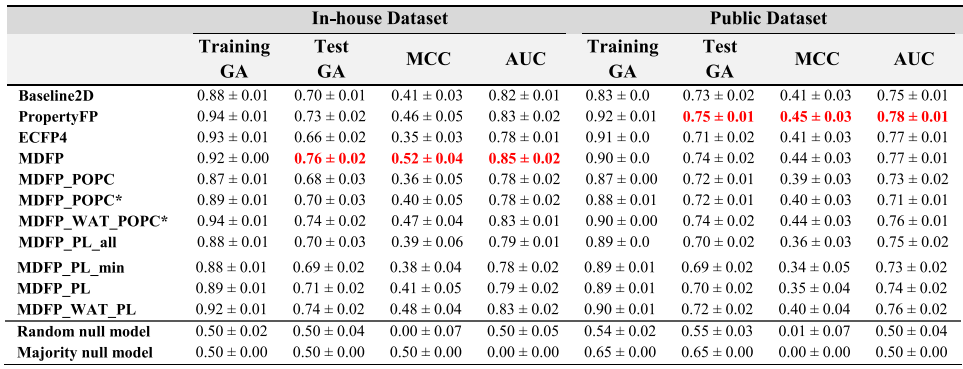
表2. 在内部数据库和公共数据库中用于RF分类器的膜-溶质和蛋白-配体MDFPs的性能概述。
图片源自JCIM.
总结
在本工作中,我们研究了MDFP/ML方法区分P-gp的底物和非底物的能力。MDFPs是根据化合物在水中的MD模拟情况来建立的,并用作四种不同的ML分类方法(即RF,GTB,SVM和meta-learner)的输入。我们证明了ML方法和指纹图谱的所有组合在最大化学多样性的化合物子集上都具有很高的相似性能。甚至仅由15个2D计数和属性组成的Baseline2D指纹也实现了高精度。对特征重要性和部分依赖性图的分析证实了先前研究的观察结果,即化合物的极性、疏水性、氢键形成能力、分子量大小和柔性与化合物成为P-gp底物的可能性相关。因此,不同的描述符表现出相似的性能,因为它们都以不同的方式对这些属性进行编码。此外,组合的ECFP4_MDFP4指纹比其单个组分表现出更高的性能,这表明基于结构的描述符和基于MD的描述符是正交的,并携带与P-gp底物分类有关的互补信息。尽管所有分类器均能很好地插值,但外部验证表明,只有在MDFPs上训练的基于树的分类器才能外推到训练集未涵盖的化学空间区域。同样,与使用其他指纹训练的模型相比,基于MDFP和PropertyFP的模型对未见化学系列化合物的泛化效果更好。
由于细胞膜和P-gp在外排转运中都起着基本作用,因此我们探索了通过将这些环境中的特征添加到化合物的MDFPs中是否可以进一步提高分类器的准确性。膜-溶质和蛋白质-配体MDFPs是通过分别在POPC脂双层和与P-gp结合的化合物的MD模拟中建立的。在这些描述符上训练的ML方法产生了良好的分类模型(准确率高于0.7)。我们发现膜-溶质和蛋白质-配体MDFPs中最相关的特征是溶质和LJ能量项的SASA,而不是代表化合物在膜中的特定行为或与蛋白质相互作用的元素。这至少部分地解释了为什么使用这些描述符并没有导致性能提高的原因。
综上所述,这些发现表明,除了描述化合物的极性、分子量大小和柔性的特征外,还需要更好地了解外排机制以及P-gp的多特异性,以指导更好的描述符的发展,从而更加准确的用于鉴定P-gp底物的计算模型。
参考文献
Combining Machine Learning and Molecular Dynamics to Predict P‑Glycoprotein Substrates, Carmen Esposito, Shuzhe Wang, Udo E. W. Lange, Frank Oellien, and Sereina Riniker. J. Chem. Inf. Model.2020, DOI: 10.1021/acs.jcim.0c00525