
引言
尽管许多潜在的药物靶标已经被发现,但是用于验证这些靶标的化学探针的开发却相对滞后。计算机虚拟筛选有望提供化学工具来解决这一问题,但长期以来一直受到假阳性率高的困扰:许多化合物相对于给定的目标蛋白结合打分排名很高,而在实验上并未显示出活性。机器学习方法目前并没有解决这个问题,本文认为这是因为没有对模型进行足够导向性的“诱饵”训练,大多数打分函数(scoring functions)在开发时对数据集的训练和测试都不够周全,因此导致模型过于简单或训练过度。作者首先建立了训练数据集(D-COID),该数据集旨在生成更具目的性的诱饵(decoy)复合物,并将这些复合物分别与可用的活性复合物匹配,通过使用该数据集进一步训练得到了基于XGBoost框架用于虚拟筛选的通用分类器(vScreenML)进而实现更有效的虚拟筛选,从而有助于在发现新的潜在药物靶标时为其提供化学探针来进行验证。
虚拟筛选方法一般可分为两类:基于配体的筛选和基于结构的筛选。基于结构的筛选通过将化学分子库的每个分子依次对接到靶标蛋白三维结构的结合口袋中,并使用打分函数来评估每种对接的蛋白质-配体复合物的结合“质量”。打分函数直观上是为了替代给定蛋白质-配体复合物的预期强度(即其结合亲和力),通过使用打分函数选择最有希望的化合物进行实验验证。因此打分函数的准确性至关重要,也是基于结构的筛选成功与否的主要决定因素。但即使是该领域的专家使用他们自己喜欢的首选方法,其中可能包括各种对接过滤器乃至人工肉眼挑选,其预测化合物中也只有约12%的化合物能够表现出活性。
当然,如果是在所筛选分子库限于含有对靶点具有天然亲和力的官能团的化合物的情况下,命中率也可能更高。相反,在没有使用其他过滤器或人工干预的情况下应用打分函数的命中率通常较低。命中率低的原因除了从建模复合物的结构中带来的不显著因素(如化合物溶解度,配体的质子化/互变异构状态建模错误等)之外,还可能归因于传统打分函数的局限性:这些函数可能包括单个能量项的参数化不足,某些可能重要的项被排除在外以及未能考虑项之间潜在的非线性相互作用。正是由于这些原因,机器学习方法特别适合开发新的在无需人工干预的情况下提高鉴定活性化合物能力的打分函数。但是进一步的分析显示,迄今为止,许多方法报告的人工基准实验中的有希望的结果可能无意中使模型过度拟合了训练数据:这可能是信息遗漏的微妙影响,当验证/测试数据并非真正与训练数据无关时,就会发生这种情况。
其他研究表明,深度学习方法的出色表现可通过检测活性化合物与诱饵化合物的化学性质之间的系统差异来实现。这些假象中的任何一种都夸大了基于基准性能的期望,但最终在后续的前瞻性评估中对这些方法进行测试时,最终导致了不可转移和令人失望的结果。
福克斯蔡斯癌症中心和堪萨斯大学的研究人员开发了结合机器学习方法的基于结构的药物筛选vScreenML,首先构建了一个新的数据集,该数据集旨在促进对机器学习模型的训练进而在现实虚拟筛选应用程序中发挥最大作用。为了建立该数据集,作者生成了一组“compelling”诱饵复合物,即一组模拟典型化合物的化合物,如果这些化合物在典型的虚拟筛选流程中能够被筛选出来,则会进入到实验测试。然后,使用此数据集来训练机器学习分类器进而将有活性的化合物从这些“compelling”的诱饵化合物中识别出来。
1. 训练数据集的生成
作者从PDB数据中挑选了一些有活性的复合物,主要包含那些具有与实际筛选过程中包含在他们的化合物库中相同的理化特性的配体分子,最终收集了1,383个活性复合物,然后对其进行能量最小化,进而避免无意中训练出仅区分晶体结构和虚拟筛选所产生的模型。接下来是得到诱饵复合物数据集,对于每种活性复合物,作者首先使用DUD-E server来识别50种具有与活性化合物相匹配的理化性质但化学结构完全不相关的化合物,然后得到每种候选诱饵化合物的低能构象,并使用ROCS针对活性化合物的3D结构进一步筛选这些构象。利用诱饵化合物与活性化合物的结构比对,作者将诱饵置于蛋白质的活性位点,并进行了与活性复合物相同的能量最小化(图1A)。作者将此数据集命名为D-COID(dataset of congruent inhibitors and decoys),并已公开提供给其他人免费使用。为了确认这种诱饵生成策略确实导致了一个具有挑战性的分类问题,作者应用了文献中报道的打分函数[nnscore,RF-Score v1,RF-Score v2,RF-Score v3,PLEClinear,PLECnn,PLECrf和RF-Score-VS]来区分D-COID数据集中的活性复合物和诱饵复合物,发现活性复合物的分数分布与诱饵复合物的分数分布有较大的重叠(图1B),这表明这些打分函数在应用于此数据集时鉴别能力有限。
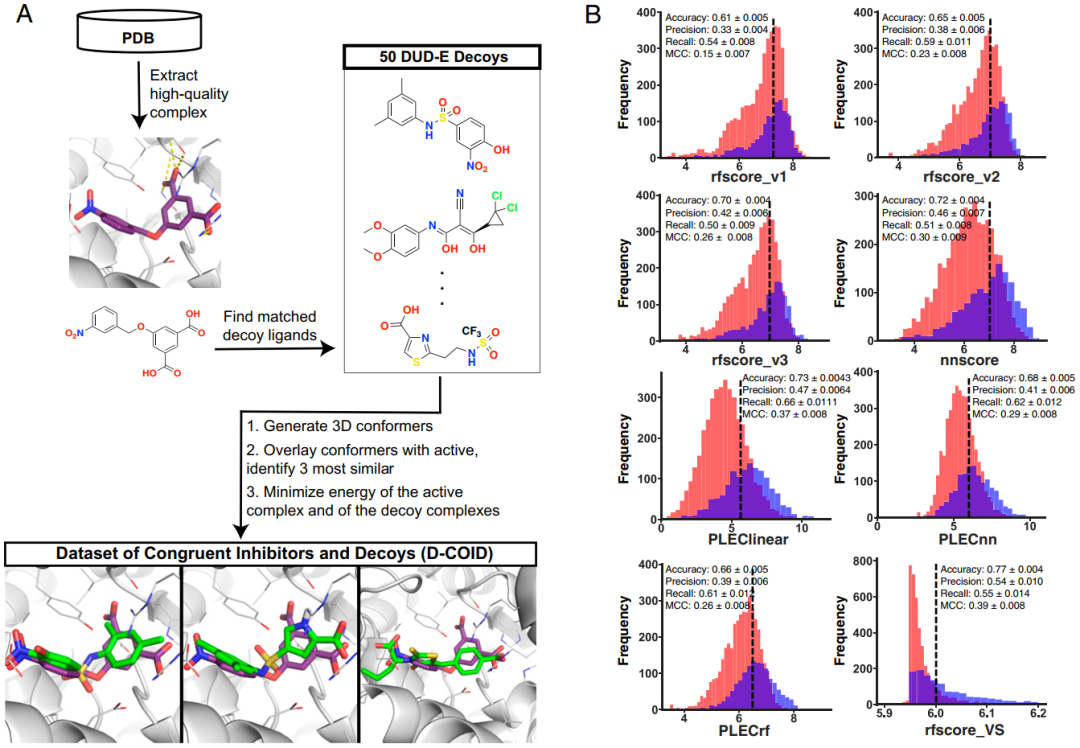
图1 D-COID训练数据集的生成
图片源于PNAS.
2. 用于识别活性复合体的分类器:vScreenML
作者以Rosetta能量函数为起点,尽管Rosetta能量函数并不常用于虚拟筛选,但这主要是因为它太慢而无法用于对接大型化合物库,在活性和诱饵复合物分类的最新基准中,Rosetta能量函数表现出与流行的FRED Chemgauss4打分函数相当的性能。此外,将Rosetta应用于D-COID集合不会产生与先前的实验(图2A)明显不同的结果,并且通过MCC (Matthews correlation coefficient)(0.40)进行了定量确认。接下来,作者使用10倍交叉验证对该打分函数中的项进行加权,以使用感知器维持Rosetta能量函数的线性函数形式,从而在D-COID分类任务中提高性能;得分的表观分离度略有改善(图2B),但MCC得到了显著改善(0.53)。这主要是因为Rosetta能量函数主要针对蛋白质而不是蛋白质-配体复合物进行了优化,并且针对特定任务重新训练其成分能量自然会提高对该任务的性能。通过使用XGBoost框架(梯度增强决策树的实现),作者观察到分配给活性/诱饵复合体的得分明显分离(图2C),MCC略有增加(0.57)。
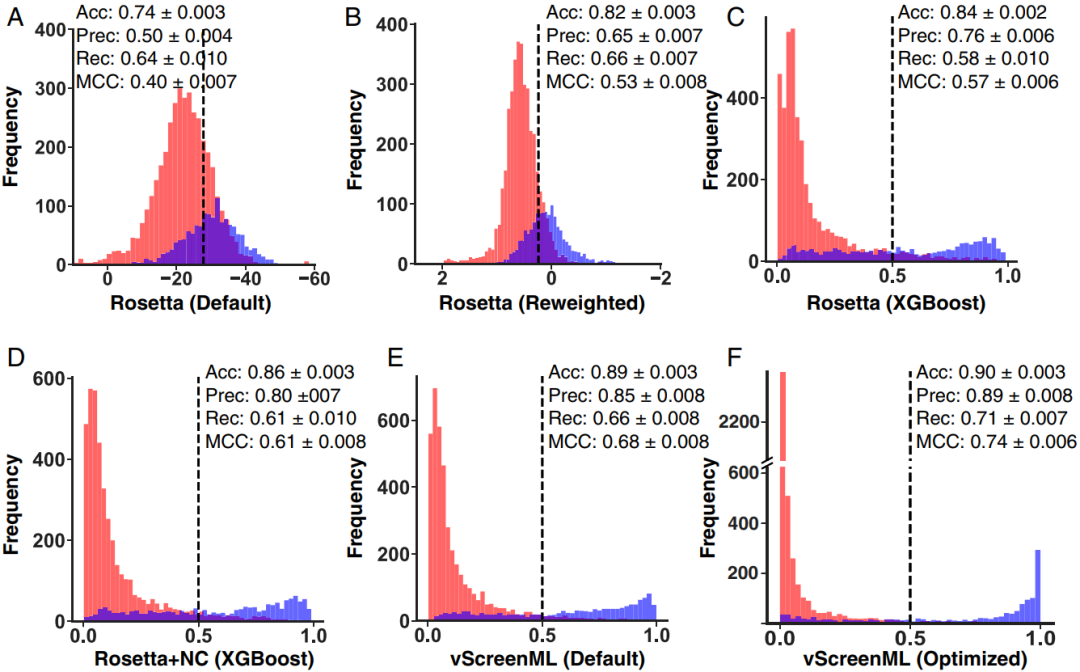
图2 vScreenML的开发。叠加的直方图显示了从D-COID对活性复合物(蓝色)和诱饵复合物(红色)进行打分时获得的得分
图片源于PNAS.
为了补充Rosetta能量函数中的现有项,作者添加了一系列由Rosetta计算的未包含在能量函数中的结构质量评估,这些项的添加产生了具有进一步提高的判别力的模型(图2D)。除此之外,作者还并入了其他结构特征,包括来自RF-Score(计算特定的成对分子间接触发生的特征),BINANA(分子间接触的分析),ChemAxon(配体特异性的分子描述符)和Szybki(捕获结合时丢失的配体构象熵的项)。通过使用该特征数据集(称为“vScreenML”)训练模型进一步增加了活性复合物和诱饵复合物之间的鉴别能力(图2E)。最后,作者使用超参数优化来进一步优化模型,并相应地开发了一个模型,该模型几乎完全分离了活性复合物和诱饵复合物(图2F)以及前所未有的MCC(0.74)。
3. 使用独立测试集对vScreenML进行基准测试。
作者针对DEKOIS数据集中的23种蛋白质中的每一种都生成30至40个活性复合物和800至1200个诱饵复合物,然后使用Rosetta对所有这些复合物进行能量最小化,将vScreenML和其他八个机器学习打分函数[nnscore,PLECnn,PLECrf,PLEClinear,RF-Score v1,RF-Score v2,RF-Score v3和RF-Score-VS]对经过能量最小化的对接好的模型进行打分并排名。为了比较不同方法之间的性能差异,作者使用一种方法(针对23个蛋白质靶标中的每一种)将EF-1%绘制为使用另一种方法的EF-1%的函数(图3A)。在这些比较中,通过将vScreenML与PLECnn(在专门针对活性复合物训练的模型中代表当前技术水平的神经网络)进行比较,PLECnn在3例(对角线上方的点)中的表现优于vScreenML,而在12例中,vScreenML表现更好(其他8例是平局)。此外,对于10个蛋白质目标中的5个,vScreenML能够将活性化合物排在前100名(即给定目标中化合物的前5%)(图3B)。除了RF-Score-VS两次达到此标准外,vScreenML在统计学上都优于所有其他替代打分函数。
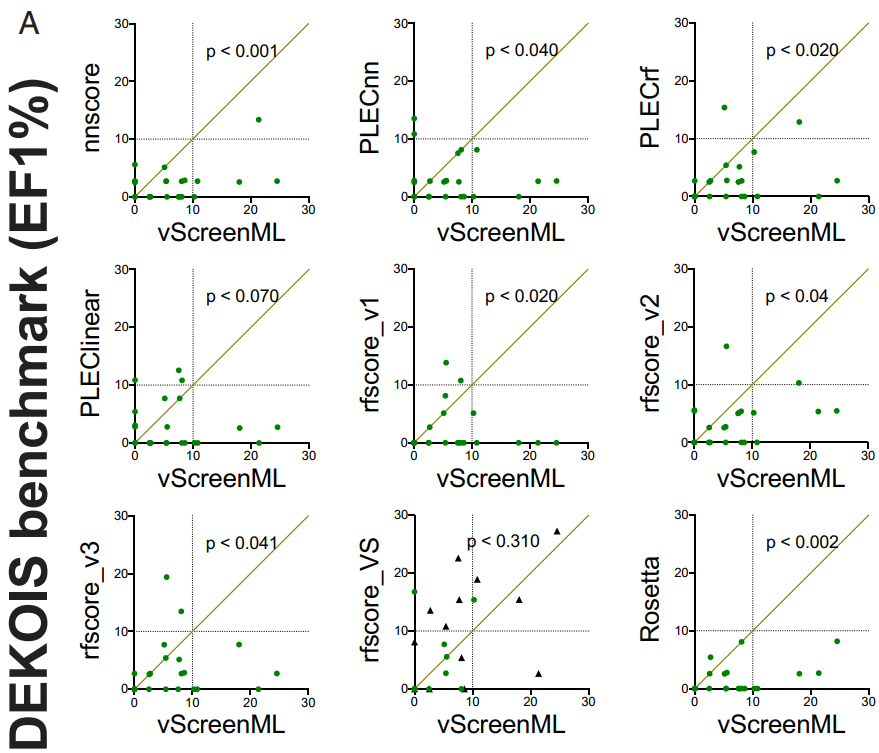
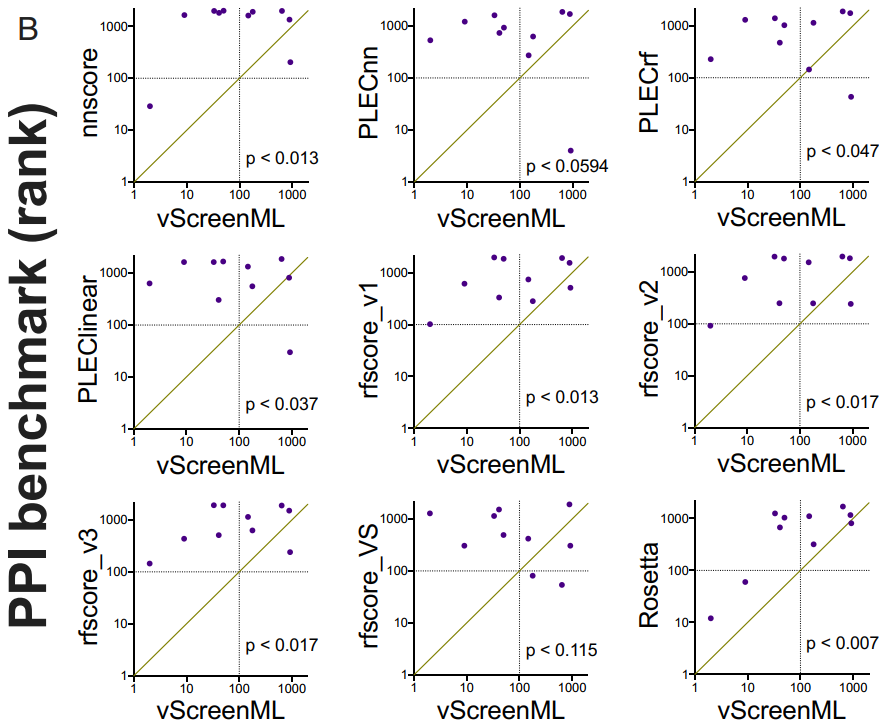
图3 使用两个独立的虚拟筛选基准,将vScreenML与其他打分函数进行比较
图片源于PNAS.
4. 在预期实验中评估vScreenML
作者选择具有代表性的人类乙酰胆碱酯酶AChE作为靶标,使用vScreenML针对一个包含7.32亿个化学分子的分子库进行了筛选,并选取了前100个作为候选分子。对于这100种初始候选化合物中的每一种,重新返回化学分子库中根据化学相似性鉴定出209个类似物;在每次搜索中将它们与母体化合物合并后,形成了一个新的包含20213种不同化合物的聚焦分子库,然后使用vScreenML对这些第二阶段候选物进行排名。将两轮筛选结果的20个得分最高的化合物综合起来,去掉彼此非常相似的化合物,并购买或合成了其余的化合物。根据标准过滤器,这些结构均未被预测为PAINS(pan-assay interference)化合物。使用比色酶测定法(图4A)以50μM的浓度测试了这23种化合物对AChE的抑制作用。可以看到vScreenML所选择的23种化合物几乎都显示出可检测到的酶抑制作用,除AC12和AC7以外,所有化合物相对于单独的二甲基亚砜(DMSO)均具有统计学上显著的AChE活性差异。在这23种化合物中,其中10种提供了超过50%的抑制作用,表明这些化合物的IC50优于50μM。此外,尽管活性最好的一对(AC6和AC3)具有广泛共享的子结构,但其仍然具有不同的化学骨架(图4B)。最后,进一步评估了最有效的抑制剂AC6的活性,在没有任何药物化学优化的情况下,该化合物的IC50为280 nM,Ki值为173 nM(图4C)。因此,应用vScreenML筛选的命中率比典型的筛选方法要高得多,并且还产生了比通常所观察到的更有效的优化起点。此外,vScreenML用于鉴定该化合物的复合物基础模型显示了广泛且几乎最佳的蛋白质-配体相互作用(图4D)。
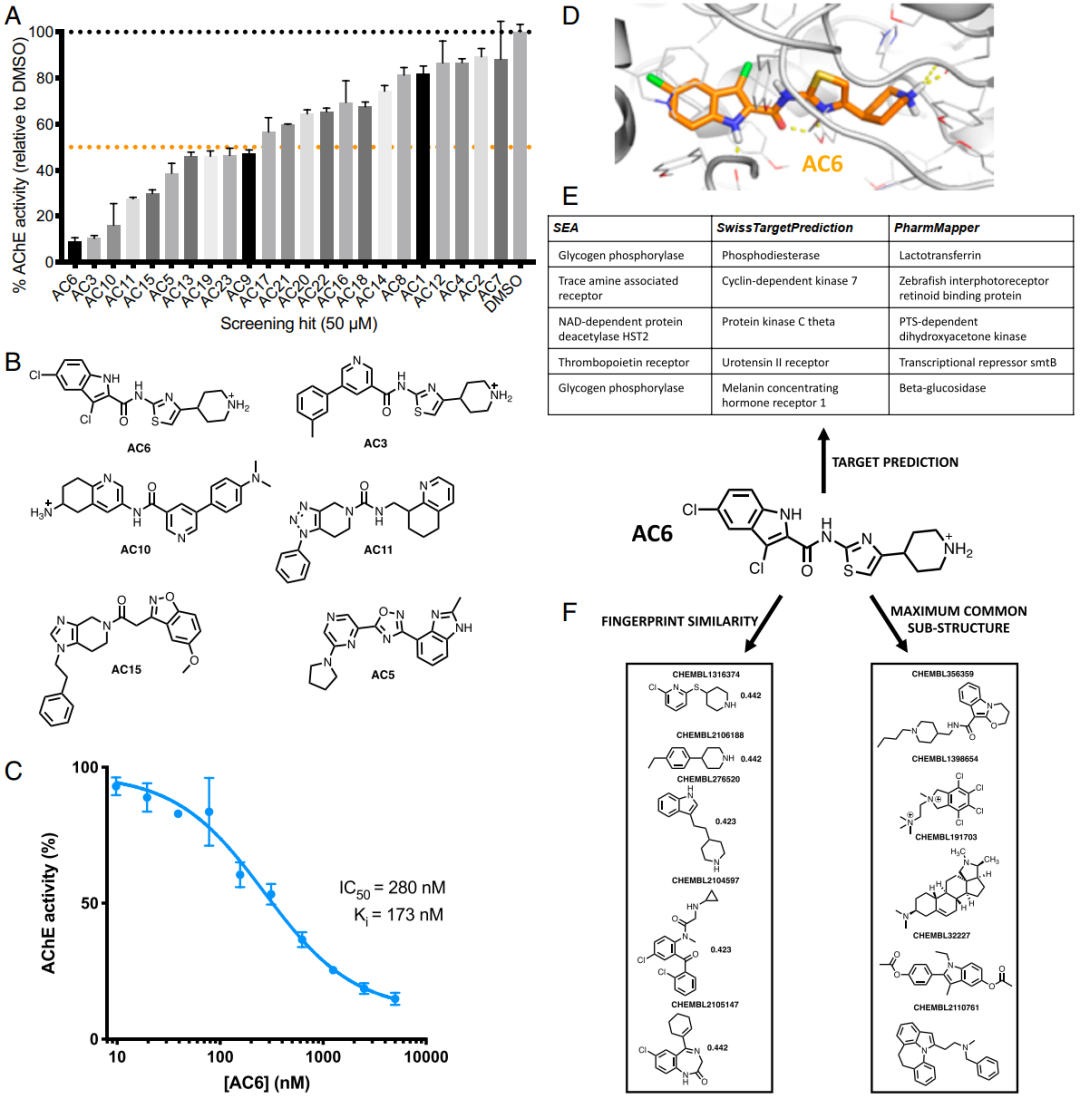
图4 在虚拟筛选中针对人乙酰胆碱酯酶(AChE)对vScreenML进行前瞻性评估
图片源于PNAS.
为了排除vScreenML在训练过程中已经“发现” AC6作为AChE抑制剂的可能性,作者进一步使用化学信息学方法来尝试找到AC6。首先为三种不同的“反向筛选”方法提供了AC6的化学结构:即相似性整合方法(SEA),SwissTargetPrediction和PharmMapper。SEA和SwissTargetPrediction根据2D相似性(即相似的化学结构)进行此搜索,而PharmMapper则评估3D相似性(即共享药效团)。对每种方法都采用了AC6的前五项预测活性,但发现这些方法中没有一个将AChE包括在其预测中(图4E)。为了直接确定迄今为止描述的与AC6最相似的AChE抑制剂,作者从ChEMBL中收集了报道具有该靶标活性的所有2,742种化合物,然后筛选了该集合以确定其与AC6的相似性(通过化学指纹或共享的子结构定义),发现通过两种方法测得的5种最相似的化合物与AC6均无明显相似性(图4F)。总而言之,这些实验证实了AC6和其他AC系列化合物在抑制AChE方面确实是新颖的化学骨架。
参考文献
Machine learning classification can reduce false positives in structure-based virtual screening.Yusuf O. Adeshina, Eric J. Deeds, John Karanicolas. PNAS. 2020, 117 (31) 18477-18488; DOI: 10.1073/pnas.2000585117