先睹为快
靶点
PPARα:过氧化物酶体增殖物激活受体α(PDB ID:3ET1和3SP6)
数据库
ChemDiv数据库(140万个小分子)
计算方法
分子对接
分子动力学模拟
计算软件
Glide,AMBER
计算流程
作者基于分子对接技术对ChemDiv数据库中140万个化合物进行了虚拟筛选,通过HTVS、SP和XP模式的级联分子对接结合肉眼筛选,命中了14个潜在的PPARα激动剂。对被命中的化合物进行了生物学评价和机理研究,最终发现了新型PPARα激动剂(化合物A-4)。作者用MD模拟揭示了化合物A-4与PPARα配体结合域结合的关键残基与稳定的结合模式,并通过突变实验进一步进行了实验验证。综上所述,化合物A-4是一种新型的先导化合物,可用于开发高效、选择性的PPARα激动剂。

虚拟筛选
考虑到不同结构类型的配体对构成结合袋的残基的取向和位置有诱导拟合效应,作者选择了PPARα的两个晶体结构(PDB ID:3ET1和3SP6)进行分子对接。对ChemDiv数据库中140万个化合物进行了虚拟筛选,使用Glide程序中HTVS(高通量)、SP(标准精度)和XP(精确精度)模式的级联分子对接依次筛选,每步筛选均只保留对接得分靠前的10%的小分子进行下一步筛选,通过考虑它们的结合姿势、蛋白质−配体相互作用和结构多样性,以及考虑它们相对于参考晶体配体的空间取向和位置进行肉眼筛选。随后对命中的14个潜在的PPARα激动剂体外生物学评价,最终发现了新的、有效的PPARα激动剂–化合物A-4(图2)。
分子动力学模拟
作者对化合物A-4与PPARα的复合物进行了100ns的分子动力学模拟。轨迹分析(包括均方根偏差(RMSD)、均方根波动(RMSF)和旋转半径(Rg)的计算)表明蛋白质−配体复合物处于平衡状态(图3和图4)。作者还考察了氢键占有率和紧密接触百分率。氢键占有率分析表明Ser280、Tyr314和Tyr464这三个关键残基与PPARα激动剂之间的氢键保持稳定,Gln277与配体之间形成了稳定的氢键,这是原始结构中没有的。在紧密接触分析中考虑了配体周围4.5Å以内的残基,结果揭示了一些密切接触比例较高的重要残基(图5),部分残基的紧密接触主要是疏水相互作用的结果。利用分子力学泊松玻尔兹曼表面积(MM/PBSA)方法,每隔50ps从平衡轨迹的最后50ns中提取一次快照,用于计算ΔGpbsa。考虑到计算时间较长,这些快照被用来计算熵贡献,间隔为10,结合自由能值ΔGbind由这两部分组成。最终得到化合物A-4的ΔGbind值为−15.4kcal/mol。作者通过结合自由能分解和丙氨酸扫描诱变(ASM)鉴定化合物A-4结合的重要残基。结合自由能分解中通过一个残基对结合自由能的贡献来评价一个残基对配体结合的重要性,其绝对值大于1.0kcal/mol被认为是重要的的残基。结果表明(图6),Thr279、Gln277、Ser280、Tyr314、Ile317、Leu321、Val324、Lys358、His440、Tyr464等残基对配体结合贡献较大,尤其是Tyr464、Tyr314和Ser280这三个残基。该结果与氢键占有率分析相一致,表明这三个氢键对配体的结合应该至关重要。另外,Gln277也有较高的结合能,表明它是一个关键残基,这也与氢键占有率分析是一致的。ASM分析结果(图7)显示,Q277A、S280A、Y314A、K358A和Y464A等突变显著改变了化合物A-4的结合自由能,表明这5个残基对化合物A-4的结合非常重要,关键残基的突变实验也证实了该结果的可靠性。
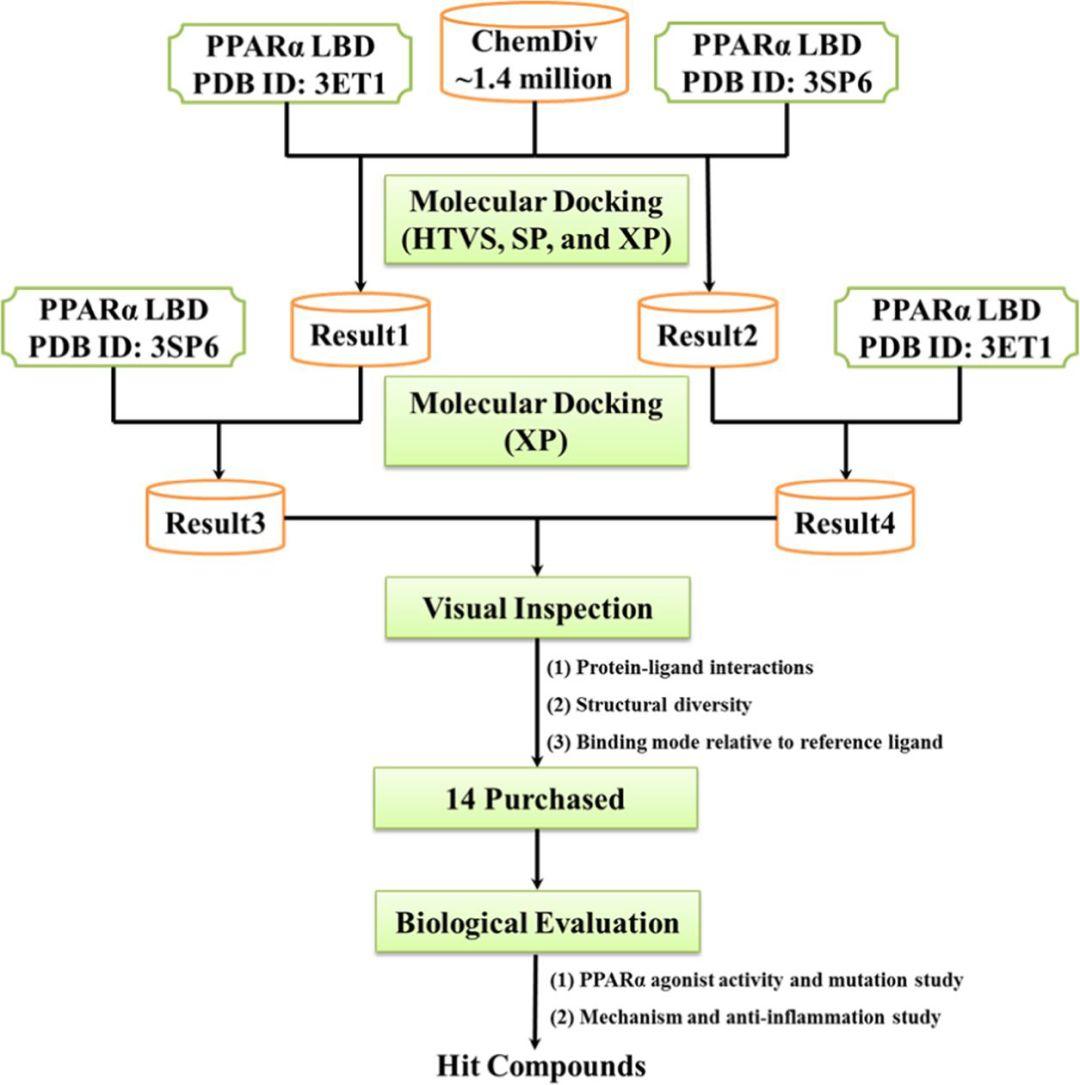
图1.虚拟筛选方法发现新型PPARα激动剂的工作流程
图片来源JCIM
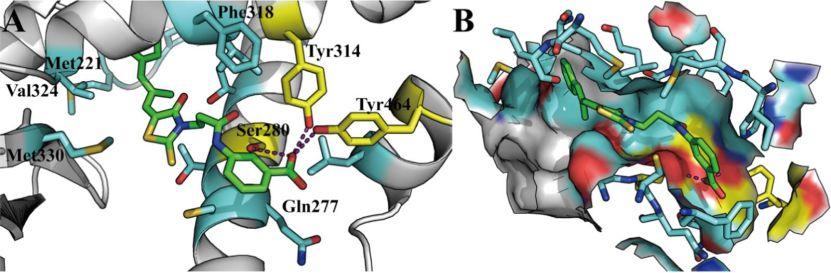
图2.分子对接预测化合物A-4与PPARα的结合模式(A)与结合构象(B)
图片来源JCIM
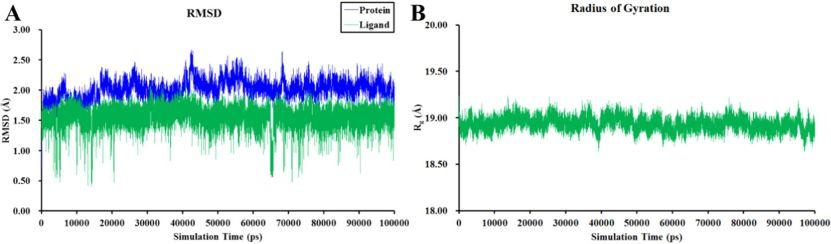
图3.分子动力学模拟中RMSD值(A)与Rg值(B)的变化
图片来源JCIM
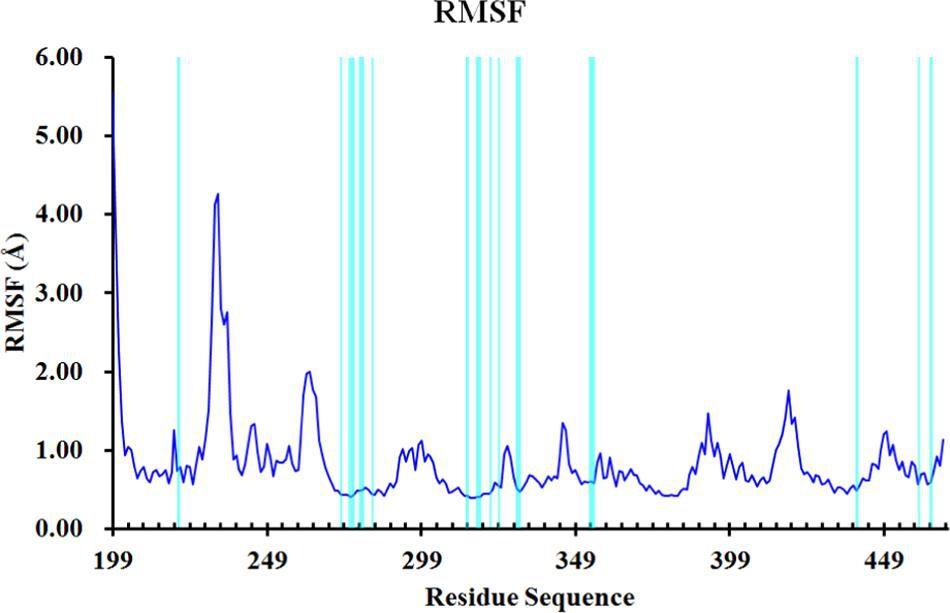
图4.蛋白质Cα原子的RMSF值计算
图片来源JCIM
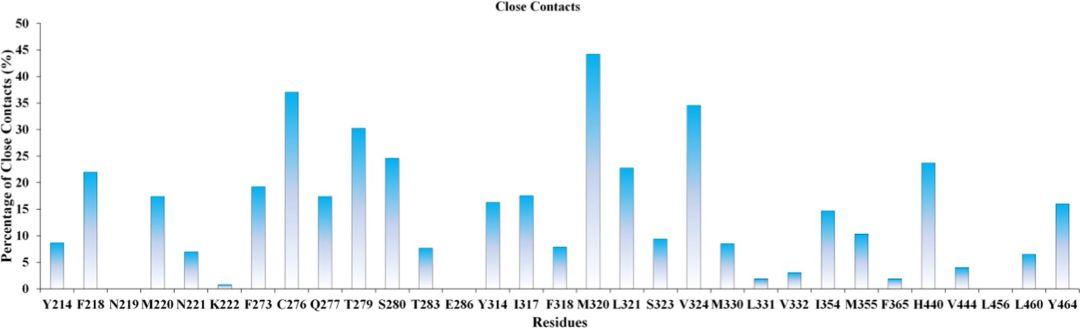
图5.PPARα-化合物A-4体系的紧密接触分析
图片来源JCIM
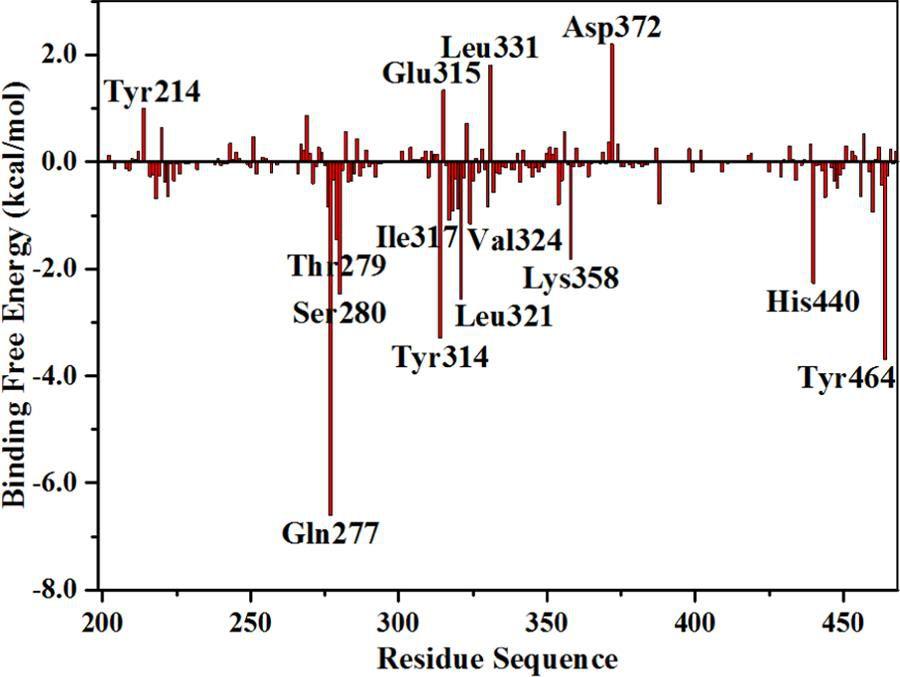
图6.结合自由能分解
图片来源JCIM
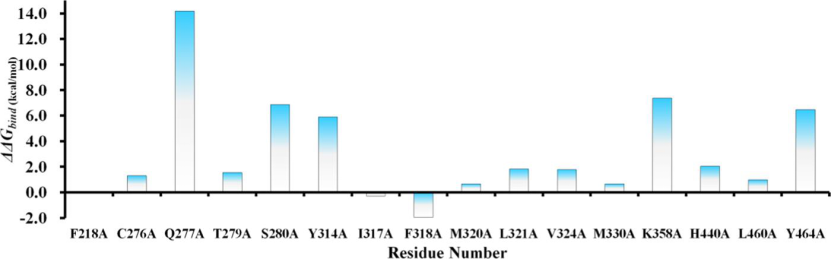
图7.PPARα-化合物A-4复合物体系的ASM分析
图片来源JCIM
参考文献:
Dai L, Feng Z, Zha R, et al. Discovery of Novel Peroxisome Proliferator-Activated Receptor α (PPARα) Agonists by Virtual Screening and Biological Evaluation[J]. Journal of Chemical Information and Modeling, 2020.DOI: 10.1021/acs.jcim.9b00838