一、生物制药中翻译后修饰(PTMs)概述
翻译后修饰(PTMs)是蛋白质生物学中至关重要的过程,指蛋白质在核糖体合成(翻译)完成后发生的化学变化。这些修饰对蛋白质的最终三维结构、稳定性及其多样化的生物功能具有决定性作用 1。PTMs极大地扩展了蛋白质的功能范围,远超其基因序列所编码的信息,从而为各种生物体带来了巨大的生物复杂性 2。目前已知的PTM类型超过650种,其中常见的修饰,如磷酸化、糖基化、乙酰化、甲基化和泛素化,占据了90%以上的已报道修饰 2。
PTMs可大致分为酶促修饰和非酶促修饰。酶促PTMs需要酶催化,由特定的酶和信号通路严格调控 5。例如,激酶催化磷酸基团的添加,甲基转移酶催化甲基的添加。非酶促PTMs则通过氨基酸侧链与活性代谢物之间的自发化学反应发生,常受细胞应激、代谢失衡和衰老等因素影响,导致不受控的修饰 5。此外,PTMs可以是可逆的,也可以是不可逆的 2。蛋白质组的这种内在复杂性和动态性,源于PTMs的庞大数量和多样化的作用机制,表明蛋白质并非静态分子,而是一个持续动态变化以适应细胞需求的系统。这种复杂性为生物制药的全面表征和质量控制带来了显著挑战,因为即使是微小的修饰变化也可能对药物的特性产生深远影响。
1.1 重组蛋白和抗体药物开发中PTM表征的独特挑战与重要性
对于生物制药产品,特别是重组蛋白和单克隆抗体(mAbs),PTMs被视为关键质量属性(CQAs),能够显著改变药物的结构、功能、稳定性以及与其他分子的相互作用 7。一个主要挑战在于重组生物治疗药物在异源表达系统(例如,非人细胞系如中国仓鼠卵巢(CHO)细胞)中生产时,可能引入与内源性人蛋白质结构不同的PTMs 1。这些结构差异可能导致患者免疫系统将药物识别为“非自身”,从而引发免疫反应并产生抗药抗体(ADAs),进而可能损害治疗效果和患者安全 1。
因此,在整个治疗性生物制药开发过程中,从早期研究到生产和储存,对PTMs进行全面表征、严格控制和持续监测是必不可少的,以确保产品的质量、疗效和安全性 1。PTMs与免疫原性之间的紧密联系,凸显了免疫原性作为生物制药关键质量属性的地位。当生物治疗药物在非人源系统中生产时,PTMs可能导致其结构异质性,使其被免疫系统识别为外来物质。这种识别直接促使抗药抗体的产生,从而可能降低药物疗效甚至引发不良反应。因此,对PTMs的表征不仅仅是理解分子结构,更是为了有效降低主要的临床风险。
1.2 蛋白质数据库(PDB)及其化学成分字典(CCD)简介
蛋白质数据库(PDB)是全球生物大分子三维结构数据的核心存储库。作为PDB的补充,化学成分字典(CCD)是一个重要的化学参考资源 10。CCD为PDB条目中包含的所有残基和小分子成分提供全面的化学描述,包括标准氨基酸、修饰氨基酸/核苷酸、小分子配体和溶剂分子 11。
CCD中的每个条目都包含详细的化学性质,如立体化学分配、标准化化学描述符(如SMILES和InChI)、系统化学名称和理想坐标 11。这种标准化对于分子实体的明确识别和分析至关重要。CCD与PDB结构数据的每周发布同步更新,确保其时效性和全面性 11。为了适应未来的增长,全球PDB(wwPDB)正在将CCD ID扩展到五位字符 14。CCD作为PTMs在结构生物学和生物制药表征中的通用标准化语言,通过为每个独特的化学实体提供一个标准化的三字符(即将扩展为五字符)字母数字代码,确保了PTM无论其通用名称或命名变体如何,都能被精确识别。这种能力对于研究人员和监管机构交叉引用数据、理解结构影响以及确保生物制药表征的一致性具有不可估量的价值。
二、抗体和蛋白类生物药物中的常见PTM修饰类型及化学变化
生物药物中的PTMs可分为酶促修饰和非酶促/化学修饰两大类,它们对药物的性质和功能产生深远影响。
2.1 酶促修饰
这些修饰是蛋白质翻译后由特定酶(如激酶、磷酸酶、蛋白酶、转移酶和连接酶)催化发生的,并受到严格调控 5。
- 糖基化 (Glycosylation)
- 化学变化: 糖基化是指碳水化合物(糖分子)通过共价键连接到蛋白质上特定氨基酸残基的过程 3。该过程由糖基转移酶催化 5。
- 类型: 主要包括N-连接糖基化(连接到天冬酰胺残基的氮原子,在IgG的Fc区天冬酰胺297位常见)和O-连接糖基化(连接到丝氨酸或苏氨酸残基的氧原子) 1。糖基化通常是永久性修饰 16。
- 与生物制药的相关性: 糖基化是治疗性单克隆抗体(mAbs)的主要PTM,深刻影响蛋白质的折叠、构象、稳定性、溶解度、定位和活性 6。糖型谱对抗体的效应功能(如补体依赖性细胞毒性(CDC)、抗体依赖性细胞介导的细胞毒性(ADCC)和抗体依赖性细胞吞噬作用(ADCP))具有关键影响 6。异常的糖基化模式会显著改变抗体介导这些免疫反应的能力 8。此外,在非人细胞系中生产可能导致添加“外来”糖残基(例如,NS0和Sp2/0细胞产生的α(1-3)半乳糖),这在人体内具有高度免疫原性 1。糖基化作为单克隆抗体异质性、免疫原性和效应功能的关键决定因素,其对单克隆抗体效应功能和结合亲和力的影响,表明PTMs不仅是蛋白质的通用修饰,更是直接影响药物预期药理作用的精细调节器。在非人源生产平台中引入的异源糖基化模式,能够直接引发免疫反应,从而将糖基化定位为单克隆抗体最关键的质量属性之一,需要贯穿整个药物生命周期进行精确控制。
- 磷酸化 (Phosphorylation)
- 化学变化: 磷酸化是指激酶将磷酸基团(PO₄³⁻)从ATP共价添加到丝氨酸、苏氨酸或酪氨酸残基的羟基上的过程 2。这是一个可逆过程,由激酶(添加)和磷酸酶(去除)催化 5。
- 与生物制药的相关性: 尽管在IgG中不如糖基化常见,但磷酸化可以发生在重链的特定位点,并调节IgG的结构构象及其与Fc受体和抗原的结合 8。在更广泛的蛋白质生物制药中,磷酸化充当分子开关,快速调节蛋白质功能、酶活性和蛋白质相互作用,在细胞信号传导、细胞周期调控和代谢中发挥重要作用 5。引入一个大的双阴离子基团可以显著改变蛋白质的电荷和相互作用 5。
- 乙酰化 (Acetylation)
- 化学变化: 乙酰化是指乙酰基(COCH₃)共价添加到蛋白质上,主要发生在赖氨酸残基的ε-氨基上 2。N-末端乙酰化(添加到蛋白质的氨基末端)也存在 5。乙酰转移酶催化添加,而脱乙酰酶将其去除 5。
- 与生物制药的相关性: 乙酰化可以影响蛋白质的折叠、稳定性和相互作用 8。对于IgG,它可能显著改变静电特性,影响结合亲和力以及与其他蛋白质或抗体的相互作用 8。除了抗体,乙酰化也是基因表达、染色质重塑和代谢的关键调节因子 5。
- 甲基化 (Methylation)
- 化学变化: 甲基化是指一个或多个甲基(-CH₃)共价添加到蛋白质上,主要发生在赖氨酸和精氨酸残基的氮原子上 2。甲基转移酶催化添加,而脱甲基酶逆转这一过程 5。
- 类型: 赖氨酸残基可以发生单甲基化、二甲基化或三甲基化。精氨酸甲基化可以是单甲基化或二甲基化,后者又可分为对称或不对称 5。
- 与生物制药的相关性: 甲基化调节蛋白质功能、细胞定位和蛋白质-蛋白质相互作用 5。在生物制药背景下,特定的甲基化模式可能影响药物-靶点结合或稳定性。
- 泛素化和类泛素化 (Ubiquitination and SUMOylation)
- 化学变化(泛素化): 泛素化是指小蛋白泛素共价连接到靶蛋白上,通常发生在赖氨酸残基上 5。这一过程涉及泛素激活酶(E1)、泛素结合酶(E2)和泛素连接酶(E3)的级联反应 5。
- 化学变化(类泛素化): 类泛素化是指小泛素样修饰物(SUMO)蛋白共价连接到赖氨酸残基上,也由E1、E2和E3酶介导 5。类泛素化是高度可逆的 5。
与生物制药的相关性: 在IgG中,泛素化主要影响抗体的周转和降解,从而影响其半衰期 8。泛素化和类泛素化在通过蛋白酶体进行蛋白质降解、细胞增殖、DNA修复和信号通路中发挥关键作用 5。
- 二硫键形成 (Disulfide Bond Formation)
- 化学变化: 蛋白质中两个半胱氨酸残基的硫醇(-SH)基团之间形成共价键,产生-S-S-连接 3。这一过程对于稳定蛋白质结构和调节蛋白质折叠至关重要 5。
- 与生物制药的相关性: 二硫键结构对于IgG分子的整体结构、稳定性和生物功能至关重要 6。二硫键形成不完全或错配可能导致各种半胱氨酸相关变体(例如,游离硫醇、硫醚形成、半胱氨酸化、三硫键等),从而影响产品均一性,并可能影响免疫原性 1。
- 蛋白水解切割 (Proteolytic Cleavage)
- 化学变化: 蛋白酶对蛋白质骨架中特定肽键的不可逆切割 2。
- 与生物制药相关的类型:
- C-末端赖氨酸截断: 在哺乳动物细胞培养中生产的单克隆抗体重链C-末端常见的剪切修饰 6。这种带正电荷的赖氨酸残基的丢失会导致正电荷减少,从而引起单克隆抗体产品的电荷异质性 6。监管机构通常要求报告C-末端赖氨酸含量 1。
- N-末端环化(焦谷氨酸形成): 由N-末端谷氨酰胺(Gln)或谷氨酸(Glu)残基重排形成焦谷氨酸(pGlu) 1。这种修饰会引入电荷异质性;谷氨酰胺转化为焦谷氨酸通常会使单克隆抗体更酸性,而谷氨酸转化为焦谷氨酸则会导致碱性偏移 6。这也被视为工艺控制不足的表现 1。
- 与生物制药的相关性: 这些修饰显著增加了生物制药产品的电荷异质性,必须对其进行表征和控制以确保产品的一致性和稳定性 6。
- 脂化 (Lipidation)
- 化学变化: 脂化是指脂质分子(例如,棕榈酸、肉豆蔻酸或异戊二烯基团)共价连接到蛋白质上 2。这一过程通常由特定的转移酶介导 5。
- 与生物制药的相关性: 脂化可以调节蛋白质功能、信号转导和膜靶向 5。虽然在循环生物制药中不常见,但它与膜相关治疗性蛋白质相关。
2.2 非酶促/化学修饰
这些PTMs通过化学反应自发发生,常受细胞应激、活性代谢物和衰老等环境因素影响 5。它们通常是不可逆的 2。
- 氧化 (Oxidation)
- 化学变化: 氧化是指氨基酸残基(特别是甲硫氨酸、半胱氨酸和酪氨酸)受活性氧物种介导的修饰 2。这一过程是非酶促且通常不可逆的 5。
- 与生物制药的相关性: 氧化可能发生在单克隆抗体开发的不同阶段,包括生产、纯化、制剂和储存 6。它能显著改变单克隆抗体的物理和生物学特性,影响其效力和稳定性 6。例如,单克隆抗体互补决定区(CDRs)内甲硫氨酸残基的氧化已被报道会降低结合亲和力并缩短分解半衰期 1。
- 脱酰胺化和异构化 (Deamidation and Isomerization)
- 化学变化: 脱酰胺化涉及从天冬酰胺(Asn)或谷氨酰胺(Gln)残基中去除酰胺基团,导致形成天冬氨酸(Asp)、异天冬氨酸(iso-Asp)或谷氨酸(Glu) 1。天冬酰胺脱酰胺化通常通过琥珀酰亚胺中间体进行,然后水解形成天冬氨酸或异天冬氨酸 17。异天冬氨酸是天冬氨酸的异构体,其侧链羧基转移到主链上 17。
- 与生物制药的相关性: 脱酰胺化和异构化在重组单克隆抗体中常见且普遍存在,对调节其异质性和稳定性具有重要作用 6。可变区内,特别是重组抗体CDRs内的脱酰胺化,已被证明会损害抗体特异性和/或结合亲和力 1。这些修饰导致电荷异质性 6。
- 糖化 (Glycation)
- 化学变化: 糖化是指还原糖(例如葡萄糖、半乳糖)通过美拉德反应非酶促地添加到氨基酸残基上 5。生物制药生产中这种PTM的主要来源是细胞培养基中存在的还原糖 6。这一过程是不可逆的 5。
- 与生物制药的相关性: 糖化会引入产品异质性,特别是影响其电荷分布 6。
- 羰基化 (Carbonylation)
- 化学变化: 羰基化是指羰基(酮或醛)添加到蛋白质侧链上,通常靶向赖氨酸、精氨酸、脯氨酸和苏氨酸残基 5。这是一种由活性氧或羰基物种诱导的不可逆非酶促修饰 5。
- 与生物制药的相关性: 羰基化可以标记蛋白质进行降解,导致功能丧失和聚集 5。它是氧化应激和蛋白质损伤的重要指标。
- S-亚硝基化 (S-Nitrosylation)
- 化学变化: 亚硝基化是指一氧化氮(NO)与半胱氨酸硫醇共价结合,形成S-亚硝基硫醇 5。这是一种基于氧化还原的修饰 5。
- 与生物制药的相关性: S-亚硝基化可以调节蛋白质稳定性、定位、功能和相互作用 5。它的存在可能会改变生物制药的治疗特性。
- 焦磷酸化 (Pyrophosphorylation)
- 化学变化: 焦磷酸化是指肌醇焦磷酸酯介导的磷酸基团添加到已存在的磷酸丝氨酸残基上 5。这种修饰是不可逆的非酶促修饰 5。
- 与生物制药的相关性: 尽管在生物制药背景下讨论较少,但焦磷酸化在各种信号通路和代谢活动中发挥作用 5。
三、PTMs对生物制药特性和疗效的影响
PTMs对生物制药的质量、安全性和疗效具有多方面的影响,这些影响贯穿药物的整个生命周期。
3.1 蛋白质结构、构象和稳定性
PTMs是蛋白质加工和成熟的基础,直接影响蛋白质的物理化学性质,包括其空间构象、空间位阻和整体稳定性 7。正确的糖基化对于维持IgG的生物活性、确保正确折叠和增强溶解度至关重要,从而防止其在血液中快速清除 6。糖基化可以通过阻碍蛋白酶结合来保护蛋白质,从而提高稳定性 9。二硫键的正确形成对于IgG分子的天然结构、稳定性和生物功能至关重要 6。异常的PTMs,例如导致聚集的修饰,可能导致蛋白质错误折叠、降解受损和清除改变,最终影响药物的完整性和保质期 1。PTMs作为高阶结构和药物完整性的直接决定因素,其对蛋白质空间构象、空间位阻和稳定性的影响,揭示了PTMs是生物制药分子架构不可或缺的一部分。PTMs的任何偏差都可能导致蛋白质错误折叠或聚集,这对于药物的生产、储存和患者安全都是关键问题。因此,对糖基化和二硫键等PTMs的精确控制,不仅仅是化学纯度的问题,更是为了确保药物维持其预期的三维结构,从而保证其长期完整性和疗效。
3.2 生物活性和效应功能
PTMs是调节蛋白质药物生物活性和特定功能的核心 7。对于单克隆抗体,糖基化,特别是Fc区的糖基化,是ADCC、CDC和ADCP等效应功能的关键决定因素 6。异常的糖基化模式会显著损害抗体触发这些关键免疫反应的能力,从而降低其治疗效果 8。其他PTMs,如磷酸化和乙酰化,可以调节IgG的结构构象,并影响其与Fc受体和抗原的结合,从而影响其整体生物活性 8。单克隆抗体互补决定区(CDRs)内甲硫氨酸残基的氧化已被报道对结合亲和力产生负面影响 1。同样,CDRs内的脱酰胺化可能会损害抗体特异性和结合亲和力 1。PTMs作为治疗作用机制的精细调节器和潜在风险,它们对ADCC、CDC、ADCP等特定效应功能以及结合亲和力的详细影响,表明PTMs不仅是通用修饰,更是直接影响药物预期药理作用的关键因素。这意味着控制PTMs对于优化药物治疗窗口和确保一致的临床结果至关重要。不受控制的PTMs可能导致药效降低甚至完全丧失预期活性。
3.3 药代动力学(PK)和半衰期
PTMs通过影响生物制药产品的稳定性、溶解度以及与负责清除的细胞受体的相互作用,对其药代动力学产生深远影响 6。例如,糖基化是决定抗体半衰期和从循环中清除率的主要因素 8。糖基化模式的变化可以通过改变抗体与肝脏和脾脏等器官中受体的结合亲和力,从而延长或缩短抗体在血液中的存在时间 8。特定甲硫氨酸残基(例如IgG1和IgG2中的M252和M428)的氧化会降低其对FcRn(新生儿Fc受体)的亲和力,导致分解半衰期缩短 1。PTMs作为药物暴露和给药方案的关键决定因素,对半衰期和清除率等药代动力学特性直接影响药物在体内的停留时间,进而影响给药频率和浓度。这在分子修饰与临床药理学之间建立了直接联系,从而影响患者的依从性和治疗成本。因此,优化药代动力学对于患者依从性和治疗效果至关重要。PTMs对药物半衰期的直接影响意味着控制这些修饰不仅仅是分析工作,更是设计安全有效生物制药给药策略的关键步骤。
3.4 免疫原性
PTMs是生物治疗药物结构异质性的重要来源,可能导致其被患者免疫系统识别为“外来物质”,从而触发抗药抗体(ADAs)的产生 1。这种“非自身”识别可能源于重组药物在非人细胞系中生产时,物种和细胞特异性PTM过程的引入 1。在制造、制剂或储存过程中引入的化学修饰(CMs)也可能产生与内源性蛋白质不同的结构特征,从而可能引发免疫反应 1。生物治疗药物的聚集形式,常因异常PTMs或蛋白质变性而加剧,尤其具有免疫原性 1。这些聚集体可被抗原呈递细胞处理,导致体液免疫反应 1。
具有显著免疫原性影响的特定PTMs包括:
- 糖基化: 非人源糖残基(例如,NS0和Sp2/0细胞产生的α(1-3)半乳糖)的添加可能导致IgG和IgE抗体的产生,引发超敏反应 1。
- 脱酰胺化: 尽管某些脱酰胺化是天然的,但重组抗体可变区内,特别是互补决定区(CDRs)内的脱酰胺化,可能会损害特异性和结合亲和力,从而可能增加免疫原性 1。
- 氧化: 甲硫氨酸残基的氧化,特别是CDRs内的氧化,会影响结合亲和力并可能导致免疫原性 1。
- 免疫复合物(IC)形成: 对于单克隆抗体,其治疗目的在于结合靶点,形成免疫复合物(ICs)。这些ICs是一种聚集体,可被具有抗原呈递能力的吞噬细胞清除,可能诱导或放大自身免疫反应,从而导致免疫原性 1。
PTM诱导的免疫原性具有多方面和复杂的性质,需要全面的风险评估。免疫原性是一个关键的安全问题,其来源是多样的。PTMs可以通过多种机制(结构变化、聚集、外来表位)诱导免疫原性,这意味着生物制药开发商必须采用多管齐下的策略进行PTM控制和免疫原性测试,从简单的表征转向全面的风险管理框架。
3.5 电荷异质性
许多PTMs显著导致抗体和蛋白质生物制药产品的电荷异质性 6。导致电荷变异的关键修饰包括:
- C-末端赖氨酸切割: 带正电荷的C-末端赖氨酸残基的丢失导致正电荷减少,从而产生更酸性的变体 6。
- N-末端环化(焦谷氨酸形成): N-末端谷氨酰胺转化为焦谷氨酸通常导致更酸性的变体,而谷氨酸的转化可能导致碱性偏移 6。
- 脱酰胺化: 天冬酰胺或谷氨酰胺分别转化为天冬氨酸或谷氨酸,引入净负电荷,导致更酸性的变体 6。
- 氧化和糖化: 这些非酶促修饰也可能改变蛋白质的电荷分布,导致异质性 6。
电荷异质性作为整体PTM控制和产品一致性的关键分析标志物,其作为各种PTMs的常见结果,意味着通过监测电荷变体,可以作为PTM谱和产品一致性的重要(尽管是间接的)指标。这是一种实用且广泛使用的质量控制分析工具。由于多种PTMs影响生物制药的电荷,电荷变体分析成为评估产品质量和批次间一致性的强大、高通量方法。它充当了各种PTMs累积效应的“指纹”,对于工艺开发和质量控制具有不可估量的价值。
四、关键PTMs的化学成分字典(CCD)标识符
4.1 CCD结构及其在识别修饰残基中的作用
化学成分字典(CCD)是蛋白质数据库(PDB)生态系统不可或缺的一部分,是PDB条目中所有成分的权威化学参考 10。它为标准和修饰氨基酸、核苷酸以及其他小分子提供标准化、明确的三字符(即将扩展为五字符)字母数字标识符(CCD ID) 11。
每个CCD条目都包含详细的化学描述,例如系统名称、同义词、化学式、分子量、立体化学分配和化学描述符(SMILES、InChI) 11。这种详细程度确保了精确的化学识别。CCD不断更新以反映已提交结构中观察到的新修饰,并且正在努力标准化原子命名并为肽残基内的蛋白质骨架和末端原子提供额外注释 22。一些旧的CCD名称也可能为了清晰度而更新 19。CCD作为PTMs化学身份与其结构背景之间的关键桥梁,其与PDB条目(包含三维结构数据)的直接关联,意味着了解PTM的CCD ID可以立即访问其精确的化学定义,更重要的是,可以探索其在蛋白质结构中的三维环境。这种能力对于理解特定修饰如何从原子层面影响蛋白质折叠、稳定性及相互作用至关重要,直接指导生物制药的设计和质量控制。对于生物制药开发而言,理解PTMs的结构后果与识别其存在同样重要。CCD提供了解锁PDB中这些结构信息的标准化密钥,从而实现合理设计、有针对性的分析方法开发以及对关键质量属性(CQAs)的更深入理解。
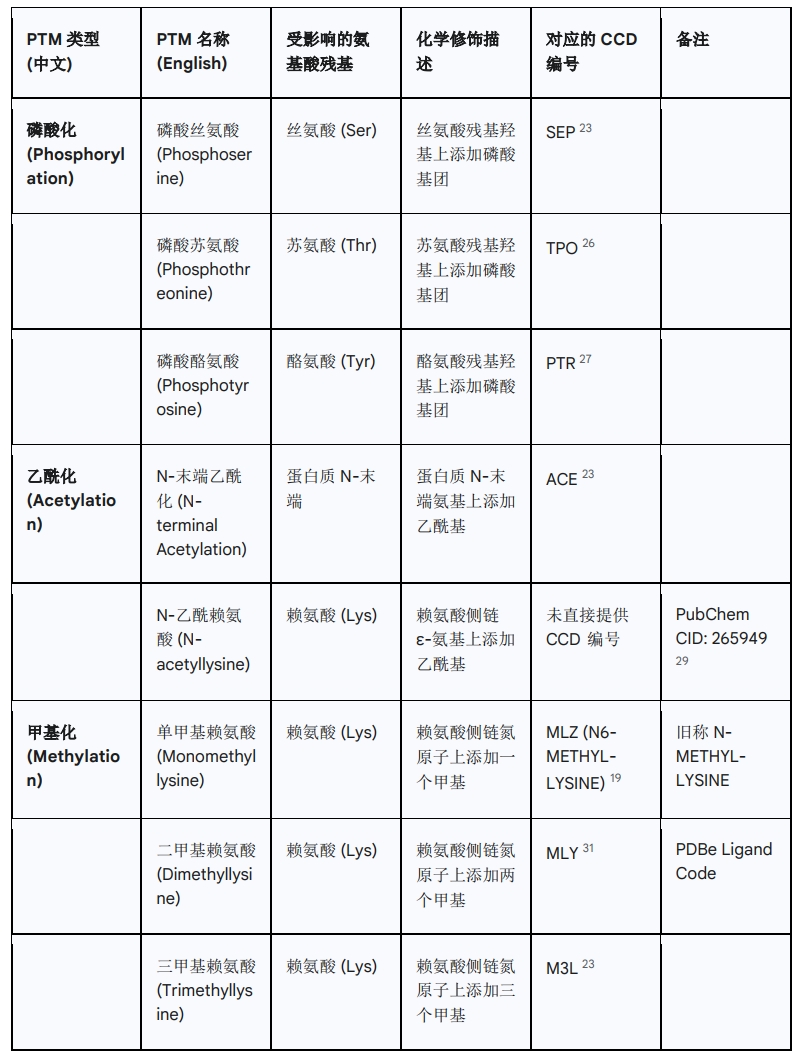
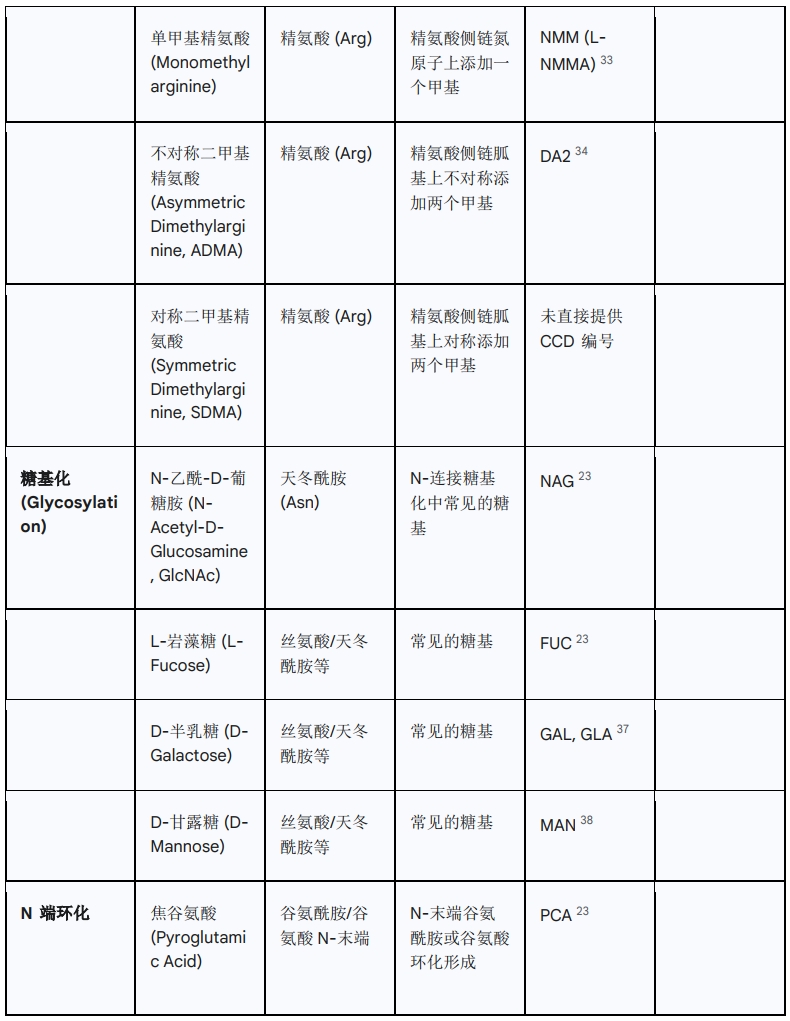
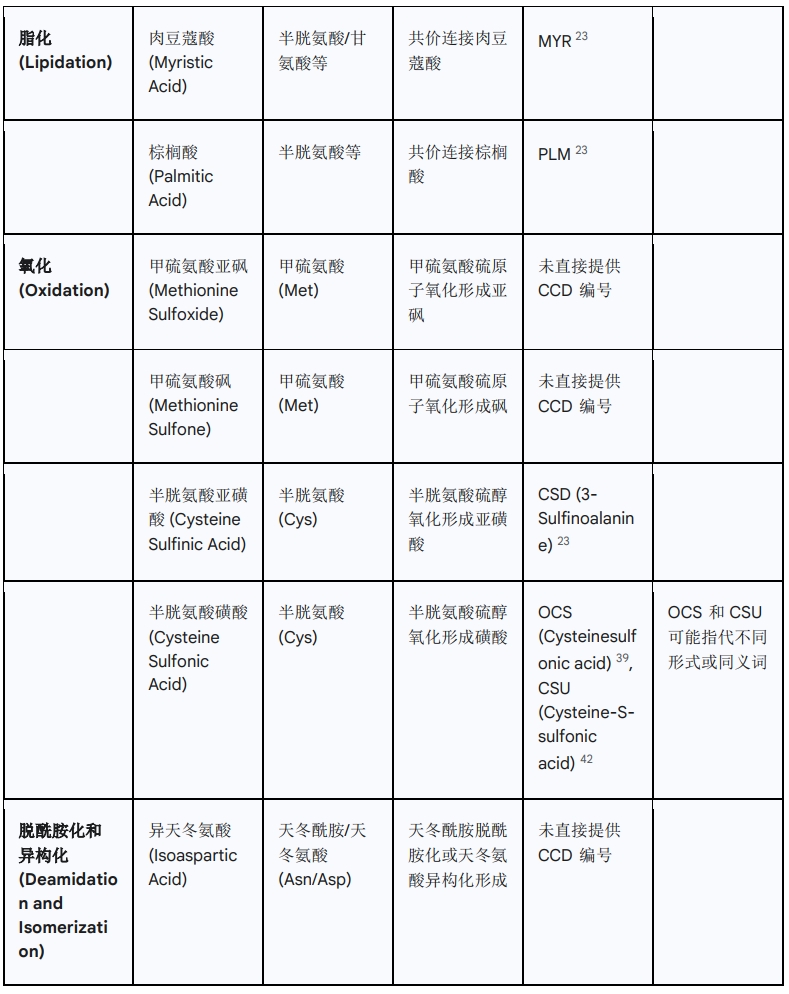
4.2 生物制药中常见PTMs及其对应的CCD标识符
下表列出了生物制药中一些常见的翻译后修饰及其在蛋白质数据库化学成分字典(PDB CCD)中的对应标识符。请注意,并非所有修饰都有直接的PDB CCD标识符,有些可能通过其母体氨基酸和修饰类型来描述,或在其他数据库(如PubChem、ChEBI)中具有标识符。对于某些复杂的或非标准修饰,可能没有单一的CCD ID。
五、结论
翻译后修饰(PTMs)是生物制药领域中蛋白质结构和功能复杂性的核心驱动因素。本报告详细阐述了PTMs的多种类型,包括酶促和非酶促修饰,并深入探讨了它们对生物制药产品质量属性的深远影响。从蛋白质的折叠、稳定性、生物活性和效应功能,到药代动力学特性和至关重要的免疫原性,PTMs在每一个环节都发挥着决定性作用。特别是对于单克隆抗体,糖基化和电荷异质性等PTMs直接影响其治疗效力和患者安全性,这凸显了在生物制药开发和生产过程中对PTMs进行全面表征、严格控制和持续监测的必要性。
化学成分字典(CCD)作为蛋白质数据库(PDB)的关键组成部分,为这些复杂的PTMs提供了一个标准化的识别和描述框架。通过为修饰残基分配唯一的CCD标识符,CCD不仅确保了PTMs的化学身份能够被精确识别,更重要的是,它将这些化学信息与蛋白质的三维结构背景紧密联系起来。这种连接对于深入理解PTMs如何影响蛋白质的分子构象、相互作用以及最终的生物功能至关重要。
综上所述,PTMs的复杂性和它们对生物制药多方面质量属性的影响,要求行业采取综合性的策略。利用CCD等标准化工具,可以促进全球范围内PTM数据的共享和互操作性,从而提高生物制药的研发效率和质量控制水平。未来的生物制药开发将继续依赖于对PTMs的深入理解和精确控制,以确保药物的有效性、安全性和一致性,最终造福患者。
引用
- Posttranslational Modifications and the Immunogenicity of …, accessed June 10, 2025, https://pmc.ncbi.nlm.nih.gov/articles/PMC4848426/
- Common Post-translational Modifications (PTMs) of Proteins: Analysis by Up-to-Date Analytical Techniques with an Emphasis on Barley | Request PDF – ResearchGate, accessed June 10, 2025, https://www.researchgate.net/publication/374455730_Common_Post-translational_Modifications_PTMs_of_Proteins_Analysis_by_Up-to-Date_Analytical_Techniques_with_an_Emphasis_on_Barley
- Post-translational Modifications | Signal Transduction – Tocris Bioscience, accessed June 10, 2025, https://www.tocris.com/cell-biology/post-translational-modifications
- Cysteine sulfinic acid and sulfinylated peptides – PMC – PubMed Central, accessed June 10, 2025, https://pmc.ncbi.nlm.nih.gov/articles/PMC12093155/
- Post-translational modifications | Abcam, accessed June 10, 2025, https://www.abcam.com/en-us/knowledge-center/cell-biology/post-translational-modifications
- Post-Translational Modifications (PTMs) Analysis – Creative Biolabs, accessed June 10, 2025, https://www.creative-biolabs.com/drug-discovery/therapeutics/post-translational-modifications-ptms-analysis.htm
- Current Technologies Unraveling the Significance of Post-Translational Modifications (PTMs) as Crucial Players in Neurodegeneration – MDPI, accessed June 10, 2025, https://www.mdpi.com/2218-273X/14/1/118
- How Post-Translational Modifications (PTMs) Impact …, accessed June 10, 2025, https://www.creative-proteomics.com/proteinseq/resource/how-ptms-impact-igg.htm
- Analysis of Post-translational Modification of Protein Drugs …, accessed June 10, 2025, https://www.creative-proteomics.com/resource/analysis-of-post-translational-modification-of-protein-drugs.htm
- ncbi.nlm.nih.gov, accessed June 10, 2025, https://pmc.ncbi.nlm.nih.gov/articles/PMC4393513/#:~:text=Summary%3A%20The%20Chemical%20Component%20Dictionary,Data%20Bank%20(PDB)%20entries.
- The chemical component dictionary | Datasets2Tools, accessed June 10, 2025, https://maayanlab.cloud/datasets2tools/landing/tool/The%20chemical%20component%20dictionary
- Chemical Component Dictionary – Bioregistry, accessed June 10, 2025, https://bioregistry.io/pdb-ccd
- wwPDB: Chemical Component Dictionary – Worldwide Protein Data Bank, accessed June 10, 2025, http://www.wwpdb.org/data/ccd
- PDB entries with extended CCD or PDB IDs will be distributed in PDBx/mmCIF format only, accessed June 10, 2025, https://www.rcsb.org/news/feature/63ff72ccc031758bf1c30ff7
- thermofisher.com, accessed June 10, 2025, https://www.thermofisher.com/us/en/home/life-science/protein-biology/protein-biology-learning-center/protein-biology-resource-library/pierce-protein-methods/overview-post-translational-modification.html#:~:text=These%20modifications%20include%20phosphorylation%2C%20glycosylation,normal%20cell%20biology%20and%20pathogenesis.
- Common Post-translational Modifications (PTMs) of Proteins: Analysis by Up-to-Date Analytical Techniques with an Emphasis on Barley – ACS Publications, accessed June 10, 2025, https://pubs.acs.org/doi/10.1021/acs.jafc.3c00886
- Isoaspartate – Wikipedia, accessed June 10, 2025, https://en.wikipedia.org/wiki/Isoaspartate
- The Isoaspartome | Fourier Transform Mass Spectrometry Lab, accessed June 10, 2025, https://www.bumc.bu.edu/ftms/research/isoaspartome/
- wwPDB:, accessed June 10, 2025, http://wwpdb-remediation.rutgers.edu/documentation/ptm-remediation
- Learn: Guide to Understanding PDB Data: Primary Sequences – PDB-101, accessed June 10, 2025, https://pdb101.rcsb.org/learn/guide-to-understanding-pdb-data/primary-sequences
- Amino Acid Code Table – GenScript, accessed June 10, 2025, https://www.genscript.com/Amino_Acid_Code.html
- Updated Annotation and Standardization of Peptide Residues – RCSB PDB, accessed June 10, 2025, https://www.rcsb.org/news/feature/64bff95ed78e004e766a9687
- protein-modification-extension/examples/examples.md at main – GitHub, accessed June 10, 2025, https://github.com/wwPDB/protein-modification-extension/blob/main/examples/examples.md
- KEGG COMPOUND: C01005 – (www.genome.jp)., accessed June 10, 2025, https://www.genome.jp/dbget-bin/www_bget?C01005
- Post-translational modifications in the Protein Data Bank – PMC, accessed June 10, 2025, https://pmc.ncbi.nlm.nih.gov/articles/PMC11394121/
- 6SUT: Crystal structure of phosphothreonine MCR-2 – RCSB PDB, accessed June 10, 2025, https://www.rcsb.org/structure/6SUT
- 3Q32: Structure of Janus kinase 2 with a pyrrolotriazine inhibitor – RCSB PDB, accessed June 10, 2025, https://www.rcsb.org/structure/3q32
- PTR Ligand Summary Page – RCSB PDB, accessed June 10, 2025, https://www.rcsb.org/ligand/PTR
- Acetyllysine – Wikipedia, accessed June 10, 2025, https://en.wikipedia.org/wiki/Acetyllysine
- 2RHY: Crystal structure of the 3-MBT repeats from human L3MBTL1 bound to monomethyl-lysine – RCSB PDB, accessed June 10, 2025, https://www.rcsb.org/structure/2rhy
- N(6),N(6)-Dimethyl-L-lysine | C8H18N2O2 | CID 193344 – PubChem, accessed June 10, 2025, https://pubchem.ncbi.nlm.nih.gov/compound/N_6_N_6_-Dimethyl-L-lysine
- M3L – Het-PDB Navi2 (Het, accessed June 10, 2025, https://hetpdbnavi.nagahama-i-bio.ac.jp/search_api/?q=M3L
- NMM Ligand Summary Page – RCSB PDB, accessed June 10, 2025, https://www.rcsb.org/ligand/NMM
- KEGG COMPOUND: C03626 – (www.genome.jp)., accessed June 10, 2025, https://www.genome.jp/dbget-bin/www_bget?cpd+C03626
- KEGG COMPOUND: C00140 – (www.genome.jp)., accessed June 10, 2025, https://www.genome.jp/dbget-bin/www_bget?C00140
- BioLiP – The Yang Zhang Lab, accessed June 10, 2025, https://zhanglab.ccmb.med.umich.edu/BioLiP/sym.cgi?code=FUC
- KEGG COMPOUND: C00124, accessed June 10, 2025, https://www.genome.jp/dbget-bin/www_bget?cpd:C00124
- MAN Ligand Summary Page – RCSB PDB, accessed June 10, 2025, https://www.rcsb.org/ligand/MAN
- OCS Ligand Summary Page – RCSB PDB, accessed June 10, 2025, https://www.rcsb.org/ligand/OCS
- OCS – Het-PDB Navi2 (Het, accessed June 10, 2025, https://hetpdbnavi.nagahama-i-bio.ac.jp/het/OCS?datatype=mol
- ChemComp-OCS: CYSTEINESULFONIC ACID – Yorodumi – Protein Data Bank Japan, accessed June 10, 2025, https://pdbj.org/emnavi/quick.php?id=OCS
- KEGG COMPOUND: C05824 – (www.genome.jp)., accessed June 10, 2025, https://www.genome.jp/dbget-bin/www_bget?cpd+C05824
- CSU Ligand Summary Page – RCSB PDB, accessed June 10, 2025, https://www.rcsb.org/ligand/CSU
- S-Sulfocysteine | C3H7NO5S2 | CID 115015 – PubChem, accessed June 10, 2025, https://pubchem.ncbi.nlm.nih.gov/compound/S-Sulfocysteine